これまでの解析で、発現変動遺伝子の算出、機能解析、さらにパスウェイ解析を通して、発現変動した遺伝子には、どのような機能の遺伝子が多いか、どの辺りのパスウェイの遺伝子が多いのか、全体的に見ました。
また、共通して変動したものが多いのか、特定のサンプルだけ変動した遺伝子が多いのかといった変動パターンは、ヒートマップで確認します。
特定の機能やパスウェイの遺伝子が、特徴的な変動パターンを示しているのであれば、それがキーになる可能性があるでしょう。そのあとは、それらの重要と考えられる遺伝子をリアルタイムPCRでチェックしたり、タンパクを免疫染色で確認したりといったバリデーションを行って、一段落となります。ここまでの結果で、論文なる場合もあるかと思います。(本当にキーになっているかどうか証明するのであれば、siRNAでノックダウンしたり、KOマウスを作成することになるでしょう。)
上流解析 (GeneMANIA)
もう少し、結果に新規性を求めるのであれば、上流解析を行うことになります。例えば、アポトーシスのパスウェイ上の遺伝子が増加していたのであれば、それを結果と考えて、それらの遺伝子を増加させている原因の遺伝子を探すというものです。
このとき、 Ingenuity Pathway Analysis (IPA) など有償のパスウェイデータベースがあれば便利です。無償のデータベースとしては、 GeneMANIA などがあります。
仮に PIK3R5, PRKACG, CASP12 遺伝子の増加が重要な結果であると考えられるとすると、それらを共通に制御する遺伝子が存在するようであれば、原因と言えるかもしれません。
GeneMANIA で、これら3つの遺伝子を検索すると、下記のようなネットワーク図が得られます。(オプションを変更して、50遺伝子まで検索しています。)
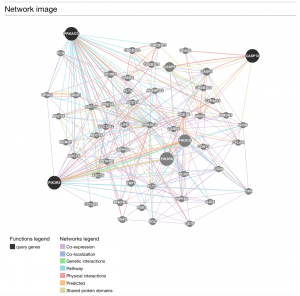
残念ながら、これら3つの遺伝子を直接共通に制御していそうな遺伝子は、見つかりませんでした。(新規性を求めるのであればということであり、必ずしもここまでの結果が要求されるわけではありません。)ネットワーク図から、次の研究のヒントは得られるかもしれません。

