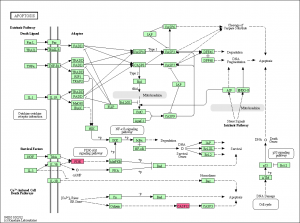
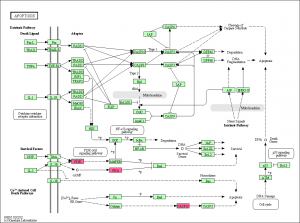
KEGG パスウェイの色付けを行う場合は、いくつか注意点があります。特に、パスウェイ上の1つのシンボルに、複数の遺伝子が含まれている状態を考慮する必要があります。
1つのシンボルに複数の遺伝子が含まれる
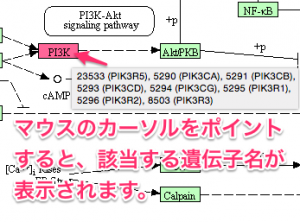
ファミリーやバリエーションのある遺伝子の場合、KEGGでは、複数の遺伝子を1つのシンボルでまとめて表記している場合があります。例えば、 PI3K などです。PI3K のシンボルには、 PIK3R5, PIK3CA, PIK3CB, … などがまとめられています。
シンボルにどの遺伝子が割り当てられているかは、各シンボルをマウスでポイントする(=カーソルを合わせてしばらく動かさない)ことで確認できます。
指定したはずの色にならない
1つのシンボルに、複数の遺伝子が割り当てられている場合、複数の色を指定したとしても、いずれかの遺伝子の色が割り当てられます。
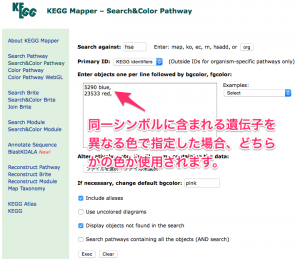
例えば、 PI3K のシンボルに含まれている 5290 (PIK3CA) を青、23533 (PIK3R5) を赤、に指定した場合、KEGG Mapper の結果において PI3K は、赤になります。 この場合、 23533 の色のみが使用されています。後に指定したほうが優先されるというわけではなく、ポイントしたときに表示される順が早い遺伝子の色を採用しているようです。(詳しくはわかりません。)
この結果、発現が下がっていたので、青く表示したかったとしても、赤に表示されるというように、指定したはずの色にならないケースがありえます。(矛盾した結果というわけではなく、ファミリーに含まれている遺伝子の変動の向きが一致しないことはしばしばあります。むしろ、全てのファミリーが同じ方向に変動したというデータを見ることが稀のように思います。)
パスウェイを発現変動遺伝子で色付けしたとしても、最終的には、エクセルに戻って、実際の ratio やシグナル値の値を確認するようにしましょう。