GEOarchive の Matrix table (マトリックステーブル) の書式を解説します。マトリックステーブルは、エクセルファイルの2枚目のシートに記入します。テンプレートに含まれる # から始まる行はコメントで、書式の説明です。”ID_REF” から始まる7行目以降の行にマイクロアレイデータを記入することになります。
ID_REF の列
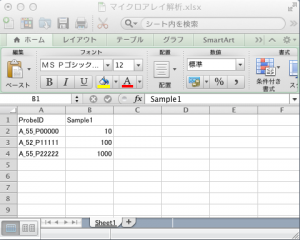
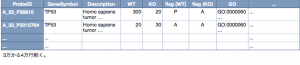
マトリックステーブルの1列目が、 “ID_REF” の列です。この “ID_REF” は、いわゆる、Probe の ID のことです。マイクロアレイのメーカーや、製品の種類によって表現が異なるので、このような表記になっているのだと思います。例えば、Affymetrix の場合は、ID, Probe Set ID (e.g. 1007_s_at)などと呼ばれます。Agilent の場合は、ID, Probe Name (e.g. A_23_P100001)のように呼ばれます。
重要なポイントは、ここで使用することになる ID を、GEO の “Platform” として登録されている情報の “ID” と一致させる必要があることです。言葉で書くと難しいのですが、通常の Probe ID (1007_s_at や A_23_P100001) を ID_REF に使用した上で、正しい Platform (GPLxxxx) をメタデータ(1枚目のシート)の platform 欄に記入していれば問題ありません。
(Illumina のマイクロアレイデータの場合、”ProbeID” と呼ばれるものが、GEO の Platform の情報においては、”Array_Address_ID” となっていることがあります。その場合は、Platform の情報における “ID” (1, 2, 3, …) に付け替える必要があります。)
SAMPLE の列
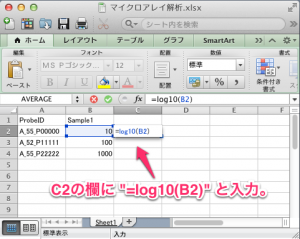
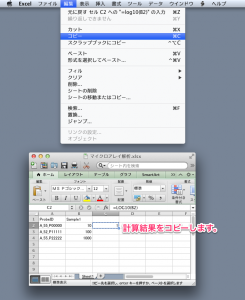
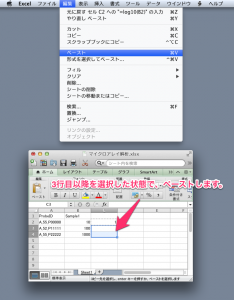
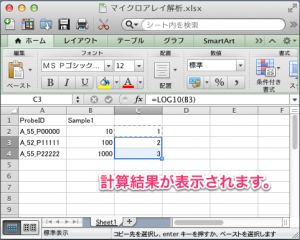
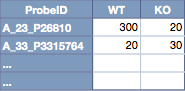
2列目以降は、サンプルの正規化されたシグナル値を記入します。1列につき、1サンプルのマイクロアレイデータということになります。もしあれば、シグナル値の次の列にフラグの値を記入します。よって、 ID_REF, SAMPLE1 のシグナル値, SAMPLE1のフラグ, SAMPLE2のシグナル値, SAMPLE2のフラグ, …, の順に続くことになります。
ここで重要なポイントは、SAMPLE1, SAMPLE2 などの各列の名前は、1枚目のシートの “Sample name” の欄に記入した名前と一致させる必要があることです。また、重複する名前も使えません。title に書いた呼び名ではありません。(間違いを防ぐために、SAMPLE1, SAMPLE2 などとしておくことをお勧めします。WT, KO などの呼び名は、title に書けます。)
フラグは、ABS_CALL (A, M, Pの情報)や、Detection p-value の情報です。各列の名前は、同じ “ABS_CALL” でもかまいません。ただし、シグナル値の列の次に来るよう、位置に気をつけましょう。