この解析例の紹介では、遺伝子発現のデータはどういうものか実感できるように、いろいろな種類のデータを取り上げたいと思います。
単純なタイムコースのデータに続き、今回は、iPS細胞のデータを取り扱います。
iPS細胞のマイクロアレイデータを取得
例のように GEO から、 iPS 細胞のマイクロアレイデータを取得します。GSE42445 のデータです。様々なバリエーションのヒトのiPS細胞 (hiPS) と幹細胞 (hES)、がん細胞 (MCF7など) のデータです。70サンプルあります。
マイクロアレイデータの正規化とボックスプロット
ここでは、rawデータを取得し、 シグナル値を取り出して、 quantile 法で正規化しました。(正規化後のデータ: Series Matrix File を取得して、そのまま用いても良いでしょう。)
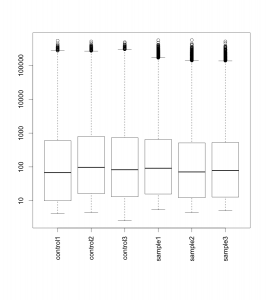
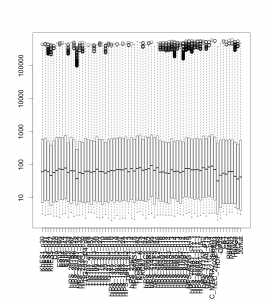
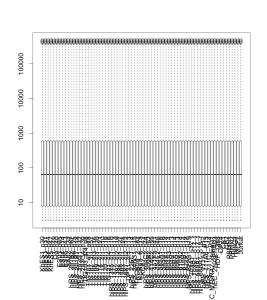
正規化前 (raw) のデータをボックスプロットで表示すると下図のようになります。特に大きく外れたサンプルはないようです。右端のがん細胞のデータが若干低いようですが、よくある程度だと思います。
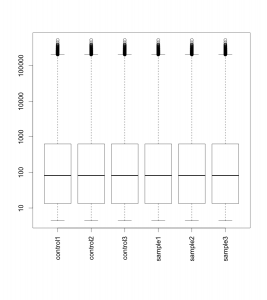
正規化後のデータをボックスプロットで表示すると下図のようになります。データの分布がそろっていることが確認できます。
参考
- Differentiation-defective phenotypes revealed by large-scale analyses of human pluripotent stem cells. Proc Natl Acad Sci U S A 2013 Dec 17;110(51):20569-74. PMID: 24259714