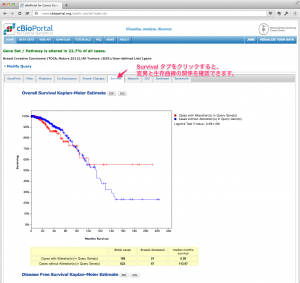
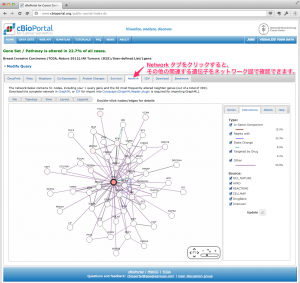
cBioPortal を利用して、特定のがん(データセット、CancerStudy)における、特定の遺伝子群の発現変動を閲覧する方法を紹介します。この操作により、自分のマイクロアレイデータで発現変動していた遺伝子が、TCGA のデータではどう変動しているかチェックすることができます。
cBioPortal のホームには、データセットを選択するための、クエリーを入力する欄があります。ここから、
データセット(がんの種類)
データ形式(変異、コピー数、マイクロアレイ、miRNA、RNASeq )
見たい遺伝子群(遺伝子の名前)
を選択します。
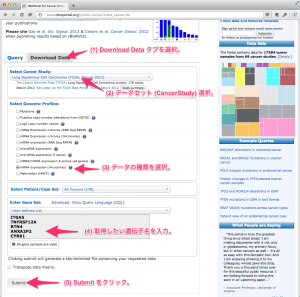
1. クエリーから、データセットを選択
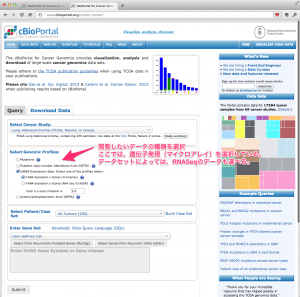
まず、データセット(がんの種類、 CancerStudy )を選択します。例では、 Lung Adenocarcinoma (TCGA, Nature, in press) を選択しています。
データセットを選択。 2. データ形式を選択
データ形式(変異、コピー数、マイクロアレイ 、miRNA、RNASeq )を選択します。選んだデータセットによって、利用可能なデータ形式のみ表示されます。すべてのデータ形式があるとは限りません。例では、mRNA =マイクロアレイのデータを選択しています。
(また、必要に応じて、データセットに含まれるサンプルのうち、特定の患者のものだけを選択することも可能。)
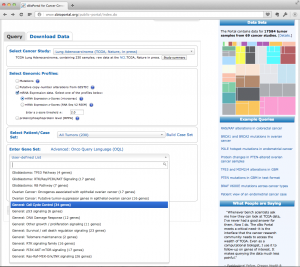
データ形式を選択。 3. 見たい遺伝子群の入力
データセットのうち、特定の遺伝子(群)を指定して閲覧できます。ボックスの中に、見たい遺伝子の Official Symbol ( 公式な遺伝子名)
遺伝子群を入力または選択する。 入力された遺伝子名が見つからない場合は、その旨が表示されます。昔の名前や通称の場合は、NCBI などで、公式な遺伝子名を確認 してください。
入力後に、 Submit をクリックします。
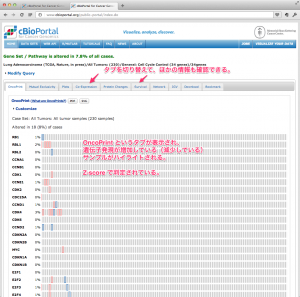
Submit 4. 結果の表示(OncoPrint)
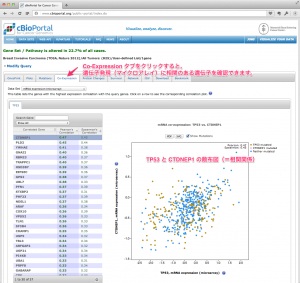
データを Submit すると、しばらくして、 OncoPrint という画面が表示されます。例では、マイクロアレイデータを選択したので、遺伝子ごとに、その遺伝子の発現が増加 している(または減少 している)サンプルがハイライトされて表示されます。ここでは、遺伝子発現の増加 、減少 が Z-score によって判定されています。ヘルプによると、 Z-score を算出するときのコントロール は、全がんサンプル の場合と、健常者のデータ の場合があるようです。どちらが使われているか、個々のデータセットを確認してください。
OncoPrint による遺伝子発現の増減を表示。