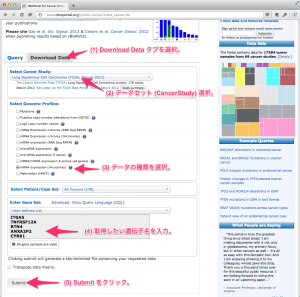
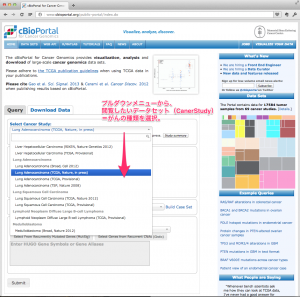
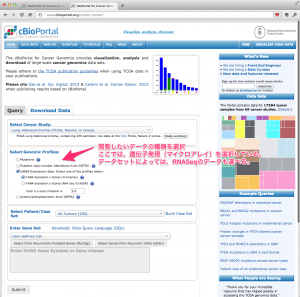
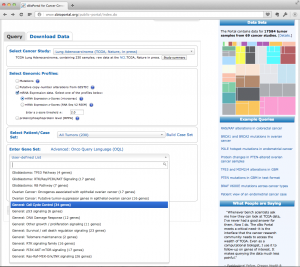
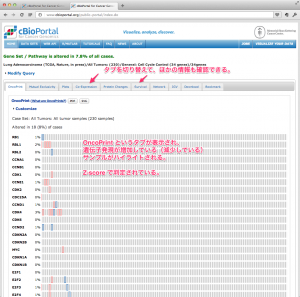
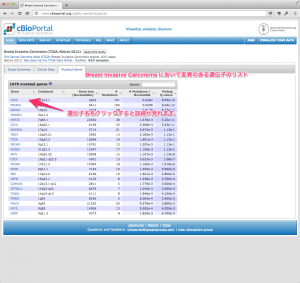
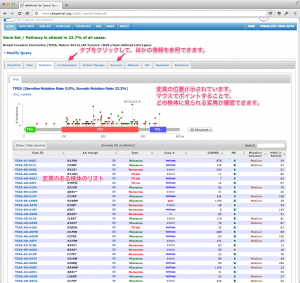
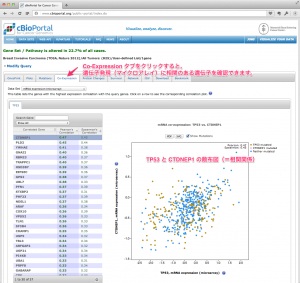
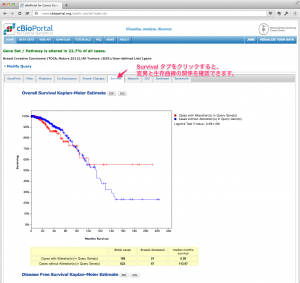
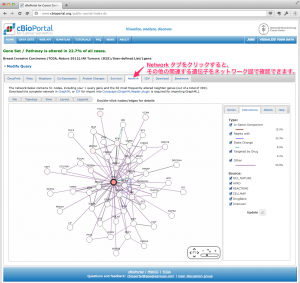
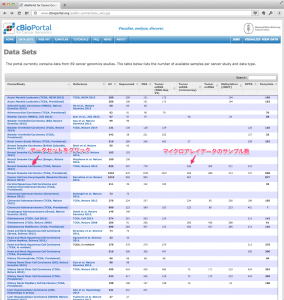
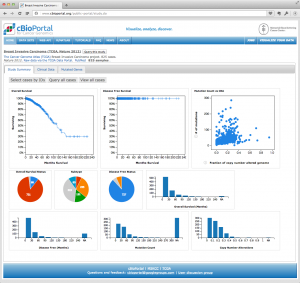
TCGA のデータを用いた研究に対する出版のガイドラインですが、2015年12月に更新があり、全てのデータセットが制限なく利用できるようになっています。
TCGA is a community resource project and data are made available rapidly after generation for community research use. In keeping with the TCGA policy of a defined moratorium on comprehensive analyses of each tumor type, moratoria on all cancer types are now lifted and all data may be used for publication without restrictions or limitations. Please review the below section on TCGA program attribution to properly acknowledge TCGA in publications and presentations; however, the additional information below concerning TCGA publication guidelines is intended for reference purposes, and need not be adhered to for any current or future uses of TCGA data.