前回に続いて、生物学的な機能で抽出した遺伝子群をクラスタリングして、ヒートマップで表示する例です。
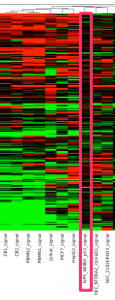
CD抗原でクラスタリング、ヒートマップ
もし、CD抗原の遺伝子をクラスタリングして、iPS細胞の株とがん細胞でクラスターが分かれるようであれば、うまく標識して区別するのに使えるかもしれません。
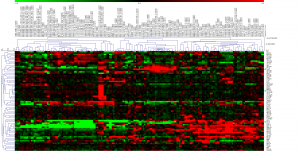
アノテーションをもとに、CD抗原の遺伝子を抽出して、クラスタリングした結果を示します。
大まかではありますが、クラスターは、iPS細胞とがん細胞で分かれるようです。しかしながら、明確にがん細胞だけに共通に発現している抗原は難しいようです。CD84などを含む一番上のクラスターでは、がん細胞においても2グループに分かれているように見えます。また、いずれの CD 抗原も iPS 細胞の株において、ところどころ赤くなっている(発現が高い)部分があります。
CD200 は、がん細胞では発現が低いように見えます。いくつかの抗原を組み合わせれば、評価できるかもしれません。(CD97の発現が高い、かつ、CD200の発現が低いなど。)
なお、マイクロアレイデータは、mRNA のレベルの結果なので、CD抗原がタンパクレベルで発現しているかどうかは、別途チェックする必要があります。
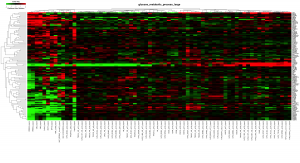
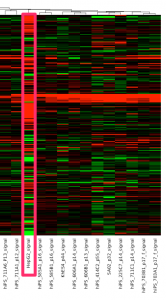
転写因子でクラスタリング、ヒートマップ
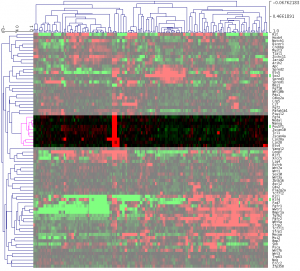
「iPS細胞とがん細胞の間で変動している遺伝子を制御している遺伝子」を探すのであれば、転写因子でクラスタリングしてみるのはどうでしょうか。

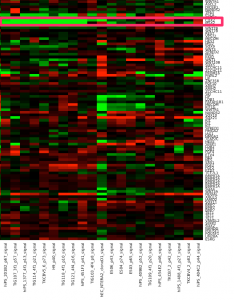
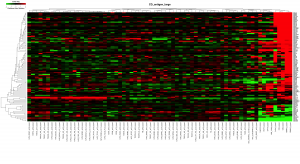
ほかの機能で抽出した遺伝子群の場合と同様に、転写因子を抽出してクラスタリングした結果も、iPS細胞とがん細胞で分かれるようです。同じ変動パターンを示すのであれば、転写を制御している可能性は示せるかもしれません。ただ、転写遺伝子の数としては、2000個程度ありますので、マイクロアレイデータだけから候補を絞り込むのは大変かもしれません。
参考
- 転写因子のヒートマップの拡大図。