Q: 発現解析(マイクロアレイ解析)で、細胞が増殖しているかどうか、判定できますか?
A: マイクロアレイデータだけから、そのサンプルが増殖状態にあるのかどうか判別するのは困難です。「増殖系に影響が出ていること」は分かります。
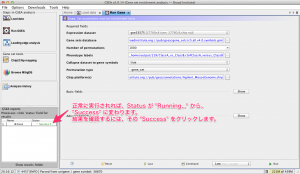
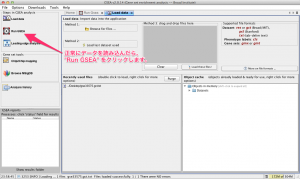
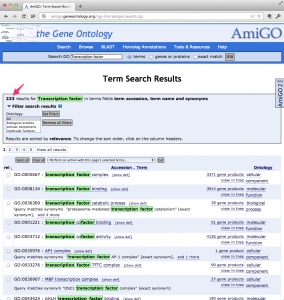
発現変動遺伝子を DAVID にかけることで、細胞周期 (Cell Cycle) 関連遺伝子が含まれているかどうかのチェックは可能です。しかしながら、こちらでも述べているように、アノテーションの問題から、単純にヒットした遺伝子の「数」だけで、結論づけることはできません。(可能性を述べるだけならできます。)
また、DAVID の結果、 Enrichment Score が 1.3 以上あり、有意と判定されたとしても、それは、増殖系の遺伝子が変動していたことを意味するだけであり、細胞数が増えているのか、減っているのかを判別するだけの効果はありません。
さらに、増殖系の遺伝子のうち、増加している遺伝子が多い=細胞が増えている、減少している遺伝子が多い=細胞が減っている、というわけでもありません。Cell Cycle 関連遺伝子といっても、細胞周期停止に関わっている遺伝子が増加すれば、細胞数は増えないでしょうし、その逆もあります。(アノテーションの問題を参照。)
細胞数が増えているかどうかを考慮するには、細胞周期だけではなく、アポトーシスの影響も考慮する必要もあるでしょう。細胞周期の遺伝子以上に、アポトーシス系が活性化されているような状況であれば、細胞数は減ることになるかもしれません。
以上のようなことから、増殖しているかどうか(増えているのか減っているのか)を判定するには、マイクロアレイデータだけではなく、フェノタイプやタンパクの状態などを確認しなければなりません。これはマイクロアレイに限った話ではないと思います。次世代シーケンサーのデータであっても同様の課題を抱えたままです。
これは、マイクロアレイデータが使えないと言っているわけではありません。「増殖状態にあるのに、機能解析の結果、Cell Cycle の遺伝子が数個しかヒットしない」という状況は考えにくいでしょう。細胞数の増減は区別できないとしても、少なくとも、「増殖系に影響が出ている」ということは、マイクロアレイデータから十分わかります。