遺伝子の変動パターンを分類するには、クラスタリングが用いられます。MeV を用いて、クラスタリングの手法の1つである k-means を使った例を紹介します。
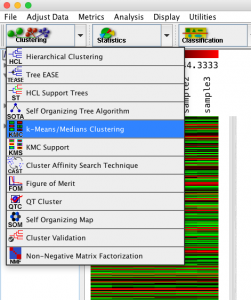
クラスタリング手法の選択
MeV でデータを読み込み、クラスタリング手法(アルゴリズム)を選択します。(ここでは、クラスタリングの前に、log2変換と中央値からの距離に直す補正を行っています。操作方法はこちら)
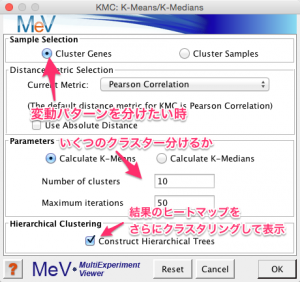
パラメーターの設定
ダイアログが表示されるので、パラメーターを設定します。ここでは、変動パターンで分類したいため、遺伝子方向のみのクラスタリングを行います。(似ているサンプルを探すのなら、サンプル方向にクラスタリングします。)
クラスターをいくつに分けるか指定します。標準では10個の設定です。
また、結果を見やすくするため、さらに階層的クラスタリングで処理するチェックを入れます。
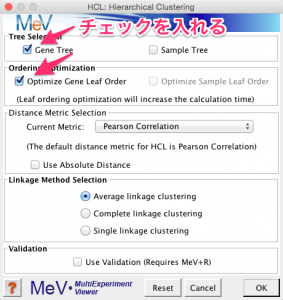
結果の表示用の階層的クラスタリングのパラメーターを設定します。同様に遺伝子方向のみ指定しています。また、最適化のオプションにチェックを入れています。ほかのパラメーターは標準設定のものを使用しました。
結果の表示
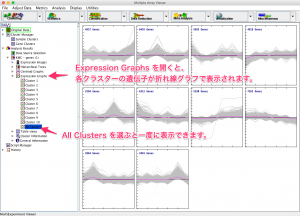
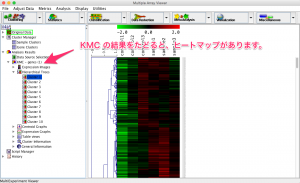
ウィンドウの左側の結果をたどると、変動パターンで10個に分けられた各クラスターのヒートマップを確認できます。
また、 Expression Graphs を選択すると、各クラスターに含まれている遺伝子の折れ線グラフが表示されます。各クラスターに含まれている遺伝子の数もここでチェックできます。