発現変動遺伝子を抽出した結果、特定のパスウェイの変動を確認するには、 KEGG パスウェイデータベースを利用できます。
KEGG のパスウェイのうち、「どのパスウェイに発現変動遺伝子が多く含まれるか」は、DAVID の結果からも確認できます。しかし、その場合は、パスウェイ上の発現変動遺伝子が☆印で示されるだけであり、増加しているのか、減少しているのかは、すぐには確認できません。(元のエクセルファイルで ratio をチェックすることになります。)
KEGG パスウェイに ratio で色付けする
KEGG には、 KEGG Mapper というサービスがあります。このサービスを使えば、特定のパスウェイの遺伝子に、増加したものは赤、減少したものは青、といった具合に色付けができます。
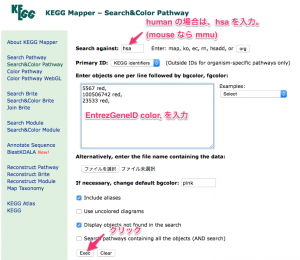
KEGG Mapper の Search against に生物種名を指定します。human なら、hsa になります。(mouse は、 mmu)
フィールドの中に、 EntrezGeneID と color をスペース区切りで入力します。複数の遺伝子に色付けする場合は、さらに , (カンマ)と改行で区切って続けます。
どの遺伝子を、どの色にするかは、発現変動遺伝子のリストを元に ratio の値を確認して、決めてください。上の例では、解析例1の 24hr の比較 (sample2/control2) において増加していた3つの遺伝子を指定しています。(この段階で、色付けしたいパスウェイの名前を指定する必要はありません。)
下部の Exec をクリックすると、検索結果の一覧が表示されます。
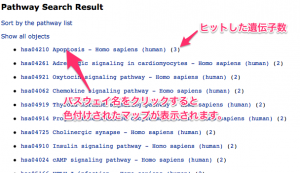
前述のフィールドに入力した遺伝子が、多くヒットしたパスウェイの順に検索結果が表示されます。さらに、パスウェイの名前をクリックして、色付けの結果を確認します。
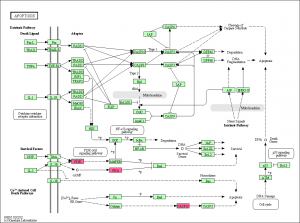
エクセルのデータから確認できるように、このパスウェイ上の遺伝子のうち、変動していたのは3つだけでした。増加していたものがありましたが、減少したものはありません。DAVIDの結果によると、Apoptosis のアノテーションを持つものは 20 個程度あるはずですが、そのうちの3遺伝子が KEGG に登録されていたことになります。(全ての遺伝子がパスウェイ上に表示されるわけではありません。)
この結果からも、アポトーシスが活性化されたというには、今ひとつ説得力に欠けるように思います(数が少ない)。CASP3 につながる CASP12 は増加していますが、 CASP3 は変動していません。ただ、 CASP3 のシグナル値を確認すると、5000程度はあるので、発現していないわけではないと考えられます。タンパクレベルで、活性化されるのであれば、mRNAの変動はなくても説明がつくのかもしれません。

「マイクロアレイデータの解析例 1.8 (KEGG パスウェイに ratio で色付けする)」への1件のフィードバック