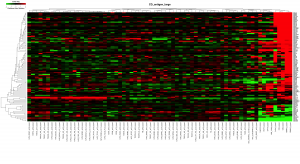
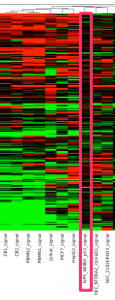
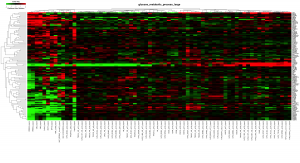
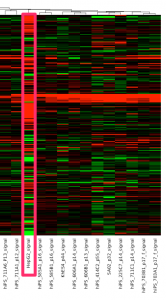
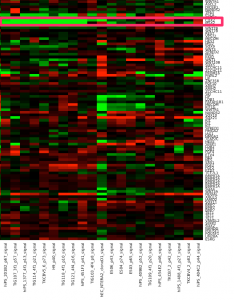
クラスタリングされたヒートマップによる表示がいつも効果的とは限りません。ヒートマップからは変動の大きさを実感しにくいと思います。散布図も確認してみましょう。
iPS細胞どうしの散布図
iPS細胞の株どうしの散布図を示します。例として、2つの株を用いています。散布図の広がり方から、変動の大きさをイメージできます。これくらいの広がり方であれば、通常、よくある程度の変動の大きさと思います。解析例1の散布図と見比べてみてください。(つまり、似ているデータではありますが、変動していない=全く同じ、というわけではないということです。)
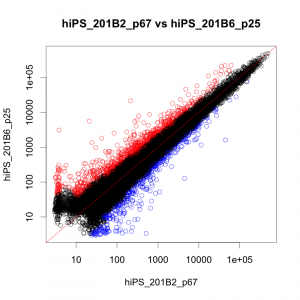
上記の散布図の色付けは、変動遺伝子を ratio と Z-score (intensity-based) の両方を用いて、判定したものになっています。
由来の異なるがん細胞どうしの散布図
では、異なる組織に由来する細胞の場合は、どれくらい広がって見えるでしょうか。HepG2 と MCF7 で比較してみましょう。
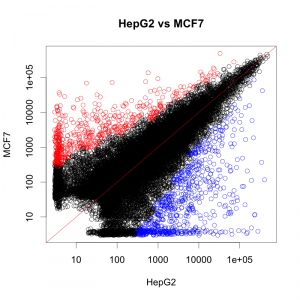
組織が異なると、散布図はこれほど広がって見えます。つまり、変動しているように見える遺伝子がそれほど多いということです。上の図では、変動遺伝子の色付けに ratio と Z-score の両方を用いているため、厳しめの判定になっています。ratio だけで判定すると、変動していると判定される遺伝子は、数千個になるでしょう。
ヒートマップでは、同じクラスターに入っていて、同じ色付けに見えるサンプルでもこれほど異なることがあります。ヒートマップだけで結果を鵜呑みにしないようにしましょう。
iPS細胞とがん細胞の散布図
同様に iPS 細胞の株と MCF7 を散布図で比較すると下図のようになります。
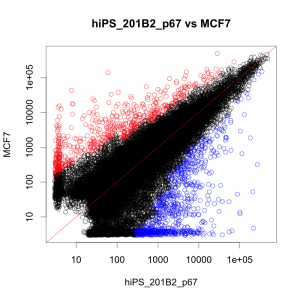
由来の異なる組織の散布図と同様に、かなり広がって見えます。遺伝子発現の状態は、大きく異なっていることが推測されます。