マイクロアレイ解析の結果、発現が変動した遺伝子のうち、転写因子を抽出するには、どうすればよいでしょうか?
発現変動遺伝子のリストがエクセルの形式であるならば、アノテーションのうち、GOを検索する方法が考えられます。
何を選ぶ?
では、転写因子を抽出するには、どの GO を選べばよいでしょうか?これは意外に難しい問題でもあります。
まず、どのような用語が GO に登録されているか、AmiGOで探してみます。AmiGOを “transcription facotr” で検索すると、223個もの用語がヒットします。
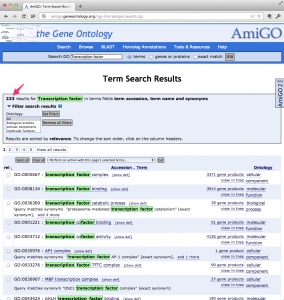
どれを選べばよいか迷ってしまうため、GOの階層構造に注目します。GOに登録された用語は、階層化されており、上位の階層に属している用語を選べば、その下位に属している用語も包括的に扱えるはずです。
上位の用語(ターム)でいいのか?
そこで、上位の階層を探してみると、上から2階層目に、 “GO:0001071 nucleic acid binding transcription factor activity” という用語が見つかります。これをアノテーションに持つ遺伝子は、 825 個(マウスの場合)あるようです。

いっけん、これでよいように思われるかもしれませんが、この用語のコメント欄を確認すると、 “Note that this term is in the subset of terms that should not be used for direct gene product annotation.” と書かれています。つまり、このような解析には、不向きな用語ということになります。
適切な用語は?
転写因子を示す用語としては、 “GO:0006351 transcription, DNA-templated” をお勧めします。名称からはピンとこないかもしれませんが、AmiGOで確認すると、この用語をアノテーションに持つ遺伝子は、2701個(マウスの場合)であることも確認できます。

このようなケースは転写因子に限りません。適切な用語を選択するには、ある程度、GOのクセや構造を理解することが必要となります。
