正しくパラメーターを設定できれば、GSEA を実行後に結果のレポートを確認できます。そのレポートの読み方を紹介します。
1. 実行状況の確認
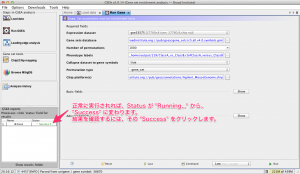
Run を押した後、ウィンドウ左下の Status には、”Running” と表示されています。Error が出ずに終了すれば、この表示が、 “Success” に変わります。(選択したリストが多くなければ、数分後には終了します。)
2. レポートの表示
“Success” の部分をクリックすると、ブラウザが起動し、解析結果のレポートが表示されます。

最初は、どこを見たらよいか分からないかもしれません。レポートの最初のページに、「炎症反応がエンリッチされた」など、結論が表示されるわけではありません。レポートは、各結果へのリンクをまとめたページです。各リンクをクリックすると、Enrichment Score (ES) の画像や、テーブル、ヒートマップなどが表示されます。
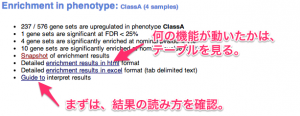
上から、ClassA (例の場合 WT)で upregulate された遺伝子群(つまり、WTで高く、KOで低い遺伝子群)のサマリー、次に ClassB (例の場合 KO)で upregulate された遺伝子群のサマリーが表示されています。
まずは、結果の見方の解説を読んでみましょう。サマリーの下に Guide へのリンクがあります。
3. テーブルの確認
とにかく、何系の遺伝子が動いたか知りたい、という場合は、テーブルを見ましょう。スコアの高い順に変動した遺伝子セットの説明(この例の場合はGO)が表示されます。”Detailed enrichment results in html format” のリンクをクリックすると確認できます。
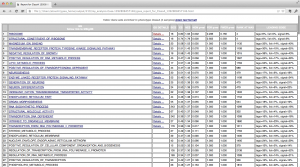
ここで指標となるのが、FDR q-val (FDR) と NOM p-val (p-value) の2つのスコアです。一般的に FDR < 25% (< 0.25) または p-value < 0.05 であれば、統計的に意味があるとされます。
これらのスコアをもとに、「免疫系の遺伝子発現が高かった」などと判断することになります。GSEAは、結論を示すものではありません。最終的な判断をするのは、あくまで、ユーザー自身です。
また、単純に生物学的な結論を下すことができないことも留意してください。
