データの読み込みができたら、GSEA を実行する際に必要なパラメーターを設定します。GSEAには、設定を変更できる非常に多くのパラメーターがあります。ここでは最低限必要なパラメーターを紹介します。
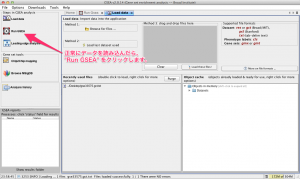
1. Run Gsea タブの表示
正常にデータを読み込んだら、左側の “Run GSEA” をクリックして、 Run Gsea タブを表示させます。
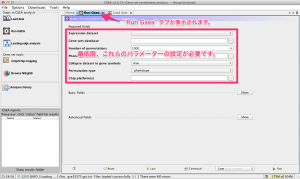
2. 最低限、設定が必要なパラメーター
Run Gsea タブに、GSEA の実行に必要なパラメーターを設定します。最低限、これらの下記の7項目の設定が必要です。(これらの設定に必要なパラメーターの多さが、GSEAを難しくしている要因の1つかと思います。)
a. Expression dataset の選択
読み込んでおいたデータセットを選択します。
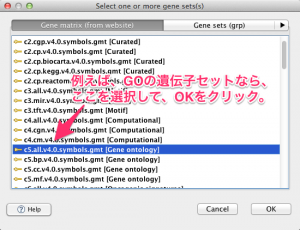
b. Gene sets database の選択
入力欄の右の “…” をクリックすると、ダイアログが表示されるので、使用する遺伝子セットを選びます。遺伝子セットは、MSigDBに登録されている遺伝子セットを利用できます。例えば、Gene Ontology (GO) の遺伝子セットを対象に GSEA を実行したいのであれば、 c5.all.v4.0.symbol.gmt を選択します。複数の遺伝子セットを一度に選択できますが、そのぶん、実行時間が長くなります。
c. Number of permutations の設定
パーミュテーションの回数を設定します。とりあえず、標準設定の1000でよいでしょう。
d. Phenotype labels
どのサンプルとどのサンプルを比較するかという情報を設定します。入力欄の右の “…” をクリックすると、設定用のダイアログが表示されます。
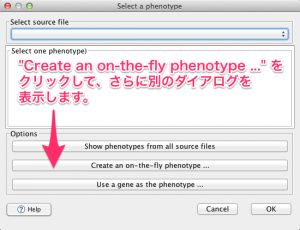
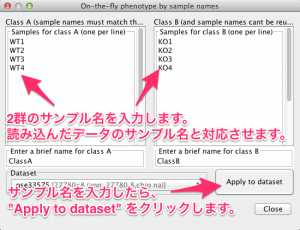
事前に設定ファイルを準備していない場合は、 “Create an on-the-fly phenotype” をクリックして、さらに別のダイアログを表示します。On-the-fly ダイアログに、比較対照となる2群のサンプル名を入力します。サンプル名は、読み込んだデータのサンプル名と対応している必要があります。サンプル名を入力したら、 “Apply to dataset” をクリックします。
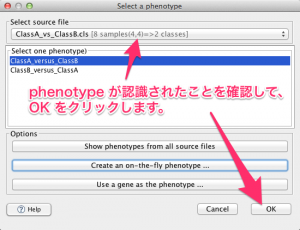
phenotype が認識されたことを確認して、OKをクリックします。
e. Collapse data set to gene symbols の設定
ProbeID (ProbeSetID) と遺伝子名を対応させる設定です。標準設定の “true” でよいでしょう。自動的に g. で設定するデータと対応が取られます。(もし、手動で行いたいのであれば、 “false” にします。)
f. Permutation type の選択
繰り返しサンプルの数が7以下 (n < 7) であれば、”gene set” が推奨されるようです。
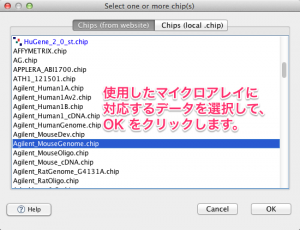
g. Chip platform の選択
用いたマイクロアレイのプラットフォーム(製品名)を選択します。ここで選んだ情報をもとに、ProbeID と遺伝子名の対応が取られます。
3. GSEA の実行
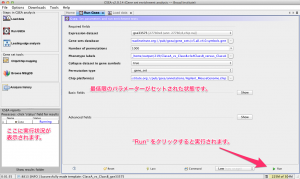
すべての設定を終えたら、ウィンドウ右下の “Run” をクリックして実行します。(ウィンドウのサイズが小さいと Run の表示が隠れて見えません。その場合は、ウィンドウの右隅をドラッグ&ドロップして、ウィンドウを広げてください。)
なお、ここでは、 繰り返しサンプルあり (n=4) の場合を紹介しました。繰り返しサンプルなし (n=1) の場合は、隠されているパラメーターの設定を変更しなければなりません。
