KEGG Mapper による色付けでは、1つのシンボルの色は、1色しか指定できません。そのため、解析例1のようなタイムコースのデータの場合、パスウェイ上での色の変化を時系列で観察するには、各タイムポイントの比較結果ごとに色付けを繰り返し行うことになります。
タイムコースデータの色付け
解析例1の場合は、16hr, 24hr, 40hr の3パターンの比較結果があるので、3回色付けを行います。前回は、24hr の結果で色付けを行っていました。このデータの場合、16hr は1つも色がつかない(変動している遺伝子が KEGG の Apoptosis にヒットしない)ので省略します。40hr の変動遺伝子を色付けした結果を下図に示します。
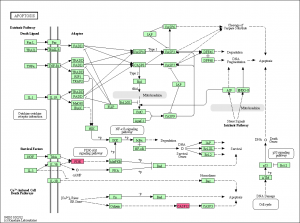
24hr の結果と比べると、PKAに色がつかなくなっています。これらの色付けされたパスウェイの図だけを見るならば、 16hr で1つも色がつかず、24hr, 40hr で CASP12 などが増加しているように見えるため、 Apoptosis が活性化しているような印象を与えるかもしれません。(閾値ぎりぎりで、変動ありとなっていない場合もありますので、正確には、エクセルなどでシグナル値をチェックする必要があります。)
KEGG WebLinks の利用
このように、複数のパターンで(or 複数のパスウェイに対して)色付けする必要がある場合は、 KEGG WebLinks を利用するのが便利です。

URL (リンク)に決まった書式(フォーマット)で、 ID とカラーを指定することで、色付けしたパスウェイの結果にアクセスできます。
今回の例であれば、下記のようなアドレスとなります。(詳しい書式は、KEGG WebLinks のページを参照してください。)
KEGG WebLinks を利用することで、直接結果にアクセスできるため、前回のようなフォームに入力する手間を省くことができます。
