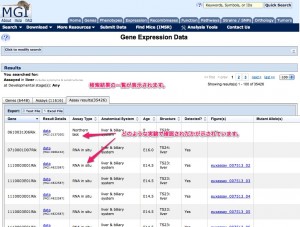
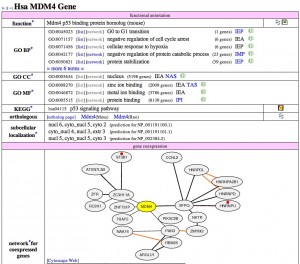
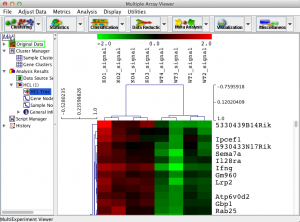
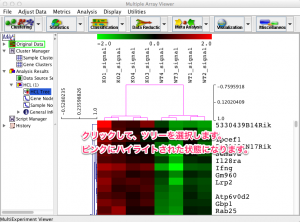
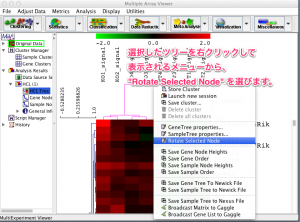
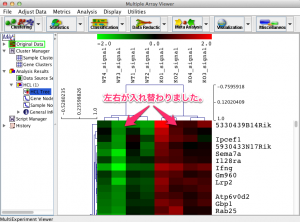
プログラミング言語の1つとして、 “R” という言語があります。”GNU R” や “R言語” とも呼ばれます。 R はプログラミング言語の中でも統計処理を得意としています。オープンソースという形で公開されていますので、誰でも無料で使用することができます。この “R” についての詳しい解説は、 RjpWiki や R-Tips を参考にされるとよいでしょう。
R のメリット
- 無料である。
- bioconductor などライブラリ(他人が用意してくれているプログラム)が多い。
- ボックスプロット、散布図、ヒートマップなどの作図もできる。
R のデメリット
- コマンドを覚える必要がある。
ボックスプロットは、エクセルでもがんばれば作成することはできます。また、t-検定も行うことは可能です。しかし、大量のサンプルを扱う場合は、コマンドで操作できる R のほうがいいこともありますし、何と言っても bioconductor の存在が大きいです。統計処理ソフトとしては、SPSS, SAS, JMP などがありますが、R であれば、 bioconductor を使ってマイクロアレイデータを比較的簡単に扱うことができます。
株式会社セルイノベーターの受託解析でも、解析ツールとして、この R と bioconductor を利用しています。