MSigDB から遺伝子セットを検索して、取得する方法を紹介します。
(1) “Search” をクリックして、遺伝子セットの検索を行います。
 遺伝子セットの検索。
遺伝子セットの検索。
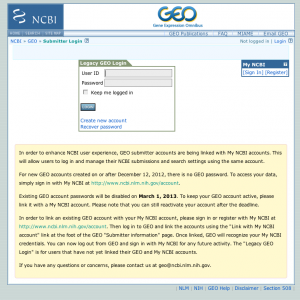
(2) メールアドレスを登録(入力)して、ログインします。
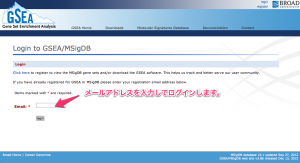 メールアドレスを入力してログイン。
メールアドレスを入力してログイン。
(3) 検索画面が表示されるので、検索したいキーワードを入力し、Search ボタンをクリックします。右側の項目を選択することで、情報の種類や生物種でフィルターをかけることもできます。
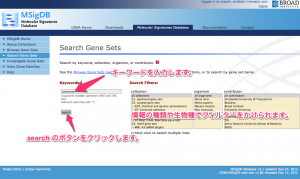 遺伝子セットの検索画面。
遺伝子セットの検索画面。
(4) 検索結果に、見つかった遺伝子セットの一覧が表示されます。リンクをクリックすることで、詳細を表示します。#genes に表示されている数字は、遺伝子セットに含まれる遺伝子の個数を意味しています。
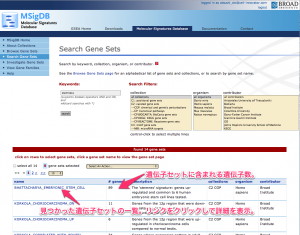 遺伝子セットの検索結果の一覧。
遺伝子セットの検索結果の一覧。
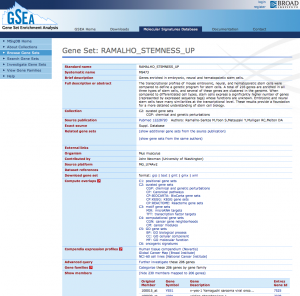
(5) 検索結果の詳細には、元になった論文の情報などが表示されます。なかほどの “Download gene set” の項目から、遺伝子セットをダウンロードできます。遺伝子セットは、複数のフォーマットで用意されています。”text”形式が最もシンプルです。この検索結果の詳細では、画面下の “Show members” の項目をクリックすることで、遺伝子セットを表示して確認することもできます。
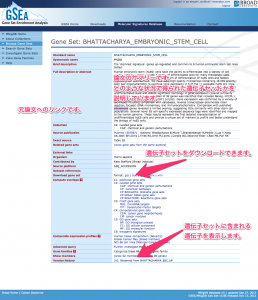 遺伝子セットの検索結果の詳細。遺伝子セットをダウンロードできる。
遺伝子セットの検索結果の詳細。遺伝子セットをダウンロードできる。
追記
なぜか、トップ画面の “Search” から検索した結果と、同画面の “Browse” から検索した結果が異なるようです(2012年12月現在)。 例は、 “stemness” で検索した結果ですが、”Search” から検索した場合がたくさんヒットしました。しかし、 “Browse” から検索しないと表示されない遺伝子セットもありました。