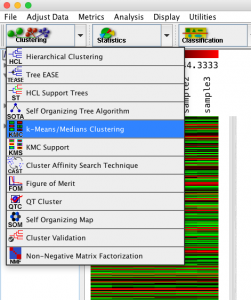
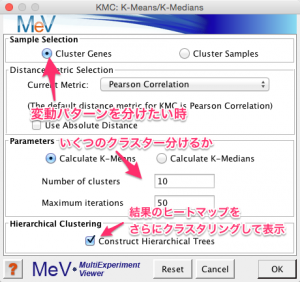
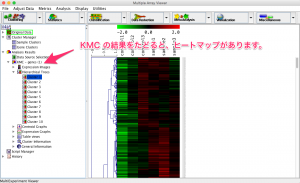
遺伝子が発現変動した結果、まず、どのような変動パターンを示す傾向にあるのか気になるのは、もっともなことだと思います。変動パターンを分類するために、クラスタリングを用いるのは正しいです。
Q: クラスタリングの結果、変動パターンは、何パターンに分類できますか?
A: 何パターンにも分類できます。
答えになっていないように思われるかもしれません。確かに、解析例1のデータは、 K-means クラスタリングの結果、10個の変動パターンに分類されました。
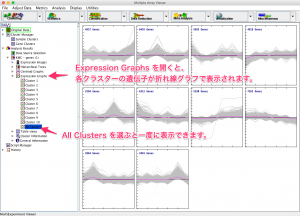
しかし、 クラスタリングの手順をよく見てみましょう。パラメーターとして、クラスターの数を入力しています。ここで「10」を設定したため、10個のクラスターが得られたのです。
例えば、このパラメーターに「27」を設定すれば、27個のクラスターに分けることができます。
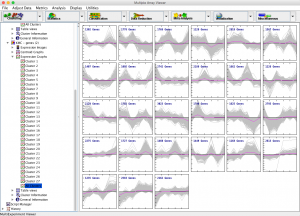
「何パターンに分けるのが適切なのか」コンピューターに決めて欲しくなるところですが、もう少しヒントがないと難しいのが現状です。(この手の問題は、コンピューターの苦手な処理です。)
参考:
K-means 法の原理については、下記のサイトの記事がイメージしやすいと思います。
> http://tech.nitoyon.com/ja/blog/2009/04/09/kmeans-visualise/