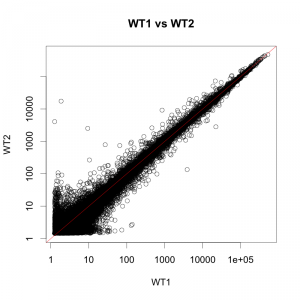
前回は、同じ条件のサンプルを散布図で比較した例を紹介しました。同じデータを MA プロットでも見てみましょう。(見方は、こちらとその続きを参照してください。)
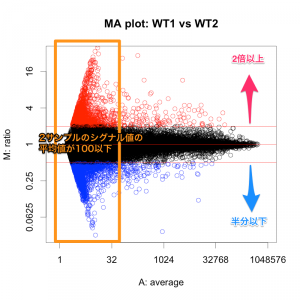
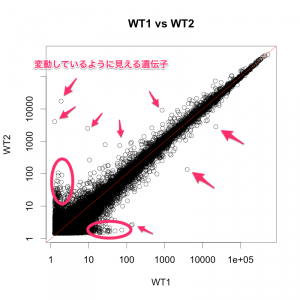
ほとんどの遺伝子が、 0.5 < ratio < 2 の区間 (-1 < M < 1 の区間) に分布しています。つまり、発現変動していないように見えます(黒い部分)。
一方、一部の遺伝子は、 ratio = 2 または、 ratio = 0.5 のライン(赤線)を超えていることも確認できます。よく見ると、これらの遺伝子は、シグナル値の低い部分に多く見られることが分かります。図では赤と青に色づけされた遺伝子が3300個ほどありますが、このうち、2サンプルのシグナル値の平均値が100以下のものが3100個ほどです。(例えば、WT1=50, WT2=100 で、ratio = 2 のものなど) 続きを読む 繰り返し実験のばらつき(2)