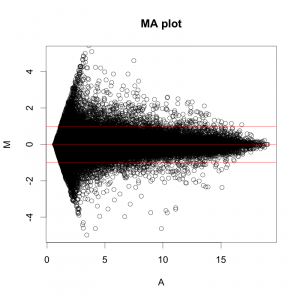
次の散布図を見てください。これは、GEOから取得した繰り返し実験を含むデータのうち、条件の同じ WT の2サンプルを比較したものです。(散布図の見方は前記事を参照。)
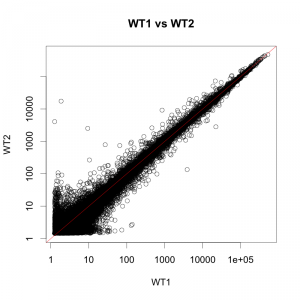
理想的に考えると、同じWTという条件で取得された遺伝子発現データなので、全く同じ結果、つまり、発現が変動している遺伝子が存在しないことが期待されるでしょう。確かに、大部分の遺伝子(4万遺伝子のうち3万以上は)変動していないように見えます。
しかしながら、変動しているように見える複数の遺伝子が、散布図から確認できます。(散布図の左上、または右下の点の存在。)
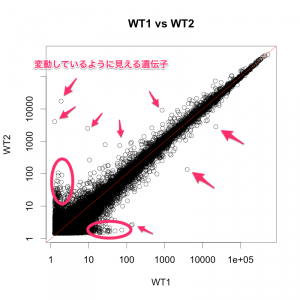
これらの遺伝子の変動は、いわゆる、「ばらつき」と考えられます。たまたま、変動したように見えただけで、次に同じ実験をしたら、観察されない変動かもしれません。
さらに、意識すべきポイントは、同じ条件であっても、たまたま変動しているように見えるものがあったということです。よって、WTとKOのように異なる条件で比較した際も、このような点が存在する可能性があるといえるでしょう。
したがって、マイクロアレイ実験の場合、1回しか実験しないことは、あまりおすすめできません。(RNA-seq も同様です。)
In particular, biological replication of each condition is crucial. –Nature Protocols, Vol.7, No.3, 2012, p569.