GEO の FIND PLATFORM の使い方を解説します。
(1) Company name: Affymetrix, Agilent, Illumina などのメーカー名を選択します。通常の製品であれば、distribution は、”commercial” のままにしておきます。
(2) 生物種を選択します。”FIND PLATFORM” のボタンをクリックして検索します。
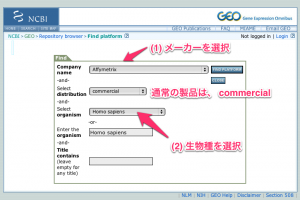
検索結果が多い場合は、Title contains の欄に、キーワードを入力して絞り込みましょう。(U133A, 1.0 Gene ST, 8x44K, 8x60K など、製品名の一部を入力します。)
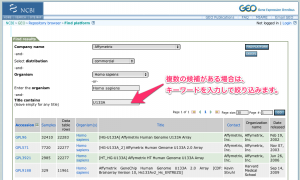
それでも、複数の候補が表示されることがあります。この場合、下記のポイントが参考になるでしょう。
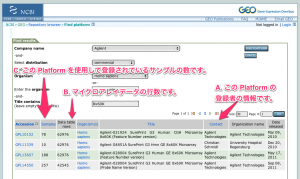
- A. 登録者の名前を見る。
- B. マイクロアレイデータの行数を見る。
- C. 登録されているサンプルの数を見る。
A. について:たいていの場合、各製品の製造元のメーカーが提供しています。個人の研究者の名前になっているものは、カスタムメイドのマイクロアレイと考えられます。
B. について:同じマイクロアレイ製品でも複数の書式で登録されている場合があります。(Agilent の Probe Name バージョンと Feature Number バージョンのように。)マトリックステーブルに入力したマイクロアレイデータの行数と同じものを選びます。(コントロールプローブの有無などで若干は異なる場合もあります。)
C. について:その Platform を使用して、GEOに登録されているサンプルの数です。これも少ない場合は、カスタムメイドのマイクロアレイのときがあります。(製品が新しいため、登録数が少ない場合もありますので、確認しましょう。)
なお、使用したマイクロアレイ製品が新しい場合は、まだ、”Platform” が登録されていないこともあります。その場合、各メーカーに問い合わせると、対応してもらえると思います。
