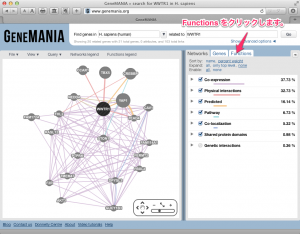
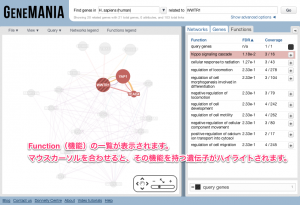
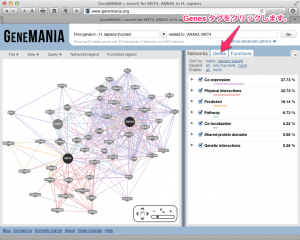
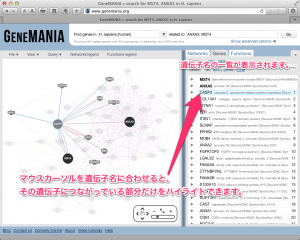
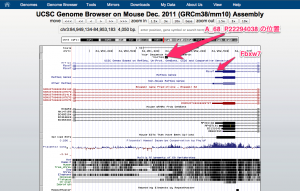
MeV を用いて、サイズの大きなヒートマップを作成した場合、結果の画像をファイルとして保存できないことがあります。
例えば、5000個の遺伝子でヒートマップを作成した場合、Set Element Size で指定する Element Width と Height を 40 x 20 くらいにしていると、この問題が発生します。
1つの対処方法としては、 Element Width と Height を 40 x 1 のようにして、縮小されたヒートマップにする方法があります。ただ、この場合、保存したヒートマップは小さすぎて、個々の遺伝子名は読めません。
数千遺伝子のヒートマップを、文字が読める大きさで画像ファイルに保存するには、MeV の利用できるメモリを増やすことで対応できるときがあります。ここでのメモリは、パソコンに搭載されたメモリのことではありません。初期状態で、パソコンに搭載されたメモリのうち、MeVの利用できるメモリは、1GBに制限されています。この制限を変更する方法を示します。(Macの場合です。)
(1) 準備(テキストエディタ)
まず、テキストファイルを編集するためのソフト(テキストエディタ)を準備してください。 “Text Wrangler” がおすすめです。必要な場合は、App Store からダウンロードしてインストールします。
 TextWrangler: App Store から入手可能。
TextWrangler: App Store から入手可能。
(2) 起動ファイル (tmev_mac.sh) の編集
次に MeV のフォルダを開きます。MeVをインストールした場所にあります。通常は、「アプリケーション」フォルダにあると思います。
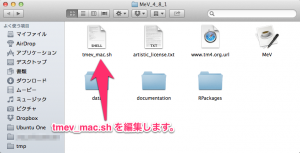 MeV のフォルダ。
MeV のフォルダ。
そのフォルダに含まれる “tmev_mac.sh” というファイルを “TextWrangler” などのテキストエディタで編集します。 “tmev_mac.sh” をクリックして、右クリックもしくは、上部のメニューから、「このアプリケーションで開く」–> 「TextWrangler」を選択します。
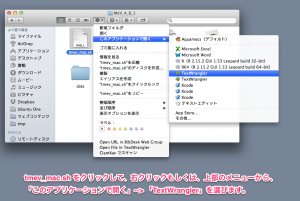 メニューから、このアプリケーションで開く–> TextWranger を選択。
メニューから、このアプリケーションで開く–> TextWranger を選択。
最終行の “1024” を “4096” などの大きい数字に変更します。ここは、パソコンに実際に搭載されたメモリの大きさと相談してください。搭載されたメモリの半分ほどは、MeVに割り当てても問題ありません。(例:メモリを8GB搭載している場合は、4GB = “4096” を指定。もし、2GB割り当てるのなら “2048” などとします。)
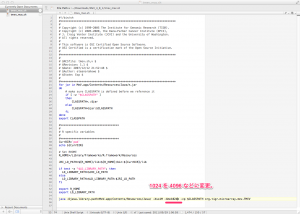 最終行を編集。1024 を 4096 など。
最終行を編集。1024 を 4096 など。
編集後は、下記のようになります。ファイルを上書き保存してください。(この設定ファイルの編集は、インストールされたMeVへの影響はありませんので、この変更により、通常の MeV が起動できくなることはありません。)
 編集後。
編集後。
(3) 編集したファイルから、MeVを起動
編集したファイルから、MeV を起動すると、メモリの制限を変更した状態で MeV を利用できます。TextWrangler のメニューの「#!」から、「Run in Terminal」を選択してください。
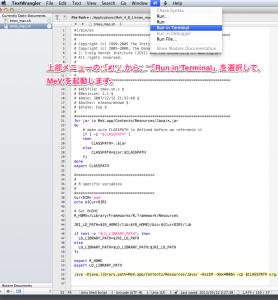 設定ファイルから MeV を起動。
設定ファイルから MeV を起動。
なお、tmev_mac.sh を上書き保存したとしても、変更したMeVを利用するには、毎回、このファイル経由で MeV を起動する必要があります。(MeV のアイコンをダブルクリックした場合は、標準状態の MeV が起動します。)