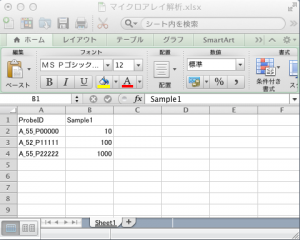
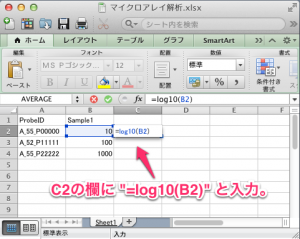
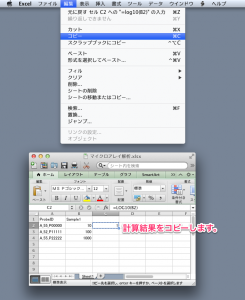
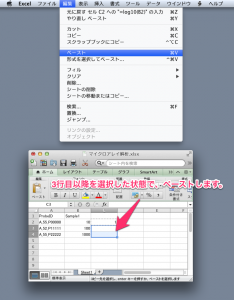
GEO からダウンロードしたエクセルファイル(テンプレート)をもとに、メタデータを記入する際、いくつか気をつけることがあります。テンプレートの記入欄の各項目のセルを矢印でポイントする(マウスで矢印を動かし、セルの上でしばらく停止させる)ことで、その注意点を読むことができます。また、テンプレートの3枚目以降のシートに記入例もありますので、そちらも参考にしてください。
シリーズ (SERIES) の欄
- title: 登録するマイクロアレイデータのタイトルです。投稿予定の論文のタイトルでもよいかもしれません。
- summary: 登録するマイクロアレイデータのサマリーです。投稿予定の論文のアブストラクトをそのまま用いても良いでしょう。
- overall design: 研究自体のデザインです。何を見る目的で、どのような条件でサンプルを取得し、どのサンプルとどのサンプルを比較したのか、記入します。
- contributor: 著者の名前です。1行に1人記入します。また、”Firstname,Lastname” のように記入します。例: Atsushi,Doi
サンプル (SAMPLES) の欄
- Sample name: サンプルの名前です。2枚目のシートのサンプル名と一致させる必要があります。重複する名前は使えません(=ユニークであることと表現されます)。テンプレートの例にあるよう、 SAMPLE1, SAMPLE2 などとしておき、WT, KO など、投稿予定の論文におけるサンプルの呼び名は、後述の title に書くほうが無難です。
- title: サンプルのタイトルです。WT, KO など、投稿予定の論文におけるサンプルの呼び名を書くことができます。
- raw data file: raw データ(生データ、正規化前のデータ)のファイル名です。Affymetrix の場合は、CEL ファイル(拡張子が .CEL のファイルです。)Agilent の場合は、 Feature Extraction から出力されるファイル(US22502696_251486831404_S01_GE1-v5_95_Feb07_1_3.txt のような名前のファイル)です。ここに書かれたファイル名のファイルをGEOarchiveのエクセルファイルといっしょにアップロードします。
- source name: 簡略化されたサンプルのコンディションを記述します。tissue, dose, timepoint, replicate1 などのように。
- organism: 生物種です。
- characteristics, tag: 特徴のタグです。tag は自由に定義できます。よく用いられるタグは、 tissue, gender, strain, age などです。特にマウスのサンプルは strain の情報を求められることがあります。列が足らない場合は、自由に追加できます。GEOにおいてデータの分類に使われるようです。
- molecule: 抽出した分子の種別です。total RNA, polyA RNA, cytoplasmic RNA, nuclear RNA, genomic DNA, protein, other から選びます。一般的なマイクロアレイでは、”total RNA”でしょう。
- label: biotin, Cy3, Cy5 など、ラベルに使用したものを記述します。
- description: ほかに記述したい情報があれば、ここに自由に記述できます。
- platform: GPL1708 など、使用したマイクロアレイのGEOにおけるIDを記入します。あらかじめ、Find Platformで調べておきましょう。カスタムのマイクロアレイを使用した場合、まず、その Platform のデータを各自が新規に登録する必要があります。そして、発行された Platform ID をここに記入します。
プロトコル (PROTOCOLS) の欄
- growth protocol: 培養時のプロトコルです。必須ではありません。
- treatment protocol: 培地などのプロトコルです。必須ではありません。
- extract protocol: RNA抽出時のプロトコルを記述します。受託解析の場合は、受託メーカーに確認することが必要になるかもしれません。
- label protocol: ラベリングのプロトコルを記述します。受託解析の場合は、受託メーカーに確認します。
- hyb protocol: ハイブリダイゼーションのプロトコルを記述します。受託解析の場合は、受託メーカーに確認します。
- scan protocol: スキャン時のプロトコルです。受託解析の場合は、受託解析の場合は、受託メーカーに確認します。
- data processing: データの前処理(正規化を含む)の内容を記述します。受託解析の場合は、受託メーカーに確認します。
- value definition: 2枚目のシートのマトリックスに含まれる値の定義です。正規化後のlog2変換された値、など。受託解析の場合は、受託メーカーに確認します。
GEOにマイクロアレイデータを登録する際には、これらの情報をほぼすべて入力する必要があります。入力が不足していれば、審査が通らず、NCBIから不足箇所の連絡があります。セルイノベーターの受託解析サービスでは、これらのメタデータの記入もお手伝いいたしますので、ご連絡ください。(このGEO登録作業のサポートも解析料金に含まれています。)