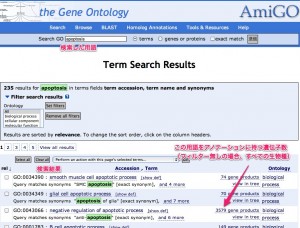
検索結果の表示と取得
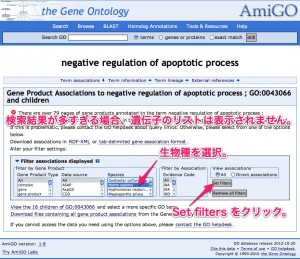
用語の検索結果の画面において、用語の右側に表示された遺伝子数をクリックすることで、その用語をアノテーションに持つ遺伝子の一覧を表示できます。このとき、遺伝子数が多い場合は一覧が表示されません。生物種を選択して、フィルターをかける必要があります。
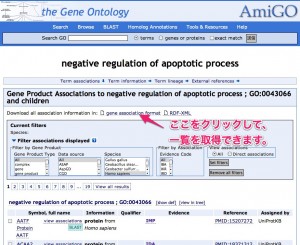
一覧が表示されます。この結果をファイルとして保存するには、 “gene association format” をクリックします。こうして取得されたリストは、マイクロアレイデータを解析する際に役立ちます。
検索結果には、選択した用語(例の場合は、GO:0043066 negative regulation of apoptotic process)と、その子供の用語をアノテーションに持つ遺伝子も含まれています。例の場合、GO:0071866 negative regulation of apoptotic process in bone marrow をアノテーションに持つ2個の遺伝子が含まれています。