初めて GEO にマイクロアレイデータを登録する際、GEO の登録要領を見ると、なかなか聞き慣れない用語ばかりがあふれているのではないかと思います。重要と思われる用語について解説します。
GEOarchive: (じーいーおー あーかいぶ)
GEOにアップロードするときのファイルの形式の1つです。1個のエクセルのファイルです。後述するメタデータとマトリックステーブルから構成されます。GEOarchive のほかに SOFTと MINiML という形式も選べますが、通常はGEOarchiveを使用した方がいいでしょう。
Deposit: (でぽじっと)
「ファイルを GEO にアップロードする作業」を指します。submit と同じような意味で使われていますが、意味としては「マイクロアレイデータをGEOに預ける」ということです。
Metadata: (めたでーた)
メタデータ、付帯情報です。著者の連絡先から、実験のデザイン、プロトコル、サンプルの組織や株の情報などを指します。GEOarchive の1枚目のシートに記述します。
Spread sheet: (すぷれっど しーと)
スプレッドシート、いわゆる、「エクセルの表」のことです。
Matrix table: (まとりっくす てーぶる)
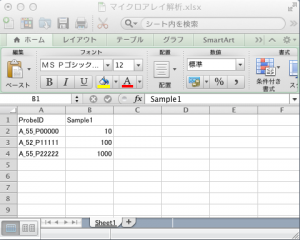
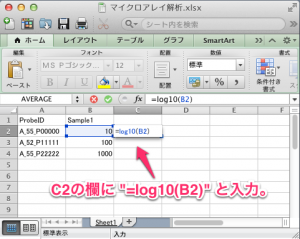
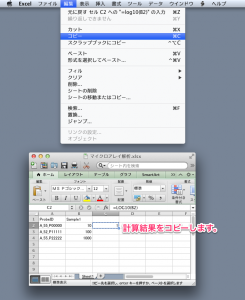
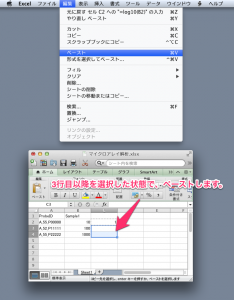
マトリックス(=行列)のテーブル(=表)です。これもスプレッドシートの1つです。中身は、マイクロアレイデータのシグナル値 (intensity) の表です。processed data または normalized data という言い方もされます。発現変動しているかどうかに関わらず、正規化後のすべてのプローブのシグナル値を含める必要があります。ratio > 2 以上の遺伝子のみという登録はできません。また、正規化に使用したサンプルをすべて含めなければなりません。4サンプルで正規化して解析したけれど、そのうち、2サンプルだけを登録するということはできません。(その場合は、2サンプルだけで正規化し直して解析しなければなりません。)GEOarchive の2枚目のシートに入力します。
Raw data files: (ろーでーた ふぁいる)
生のデータ、つまり、正規化前のデータのことです。スキャナで読み取った後に出力されるファイルです。各メーカーによって、形式が異なります。Affymetrix の場合は、CEL ファイル(拡張子が .CEL のファイルです。)Agilent の場合は、 Feature Extraction から出力されるファイル(US22502696_251486831404_S01_GE1-v5_95_Feb07_1_3.txt のような名前のファイル)です。
Platform: (ぷらっとふぉーむ)
マイクロアレイ実験に使用したマイクロアレイ(チップ)の製品の情報、またはGEOにおけるIDです。各メーカーや研究者によって、製品ごとのプローブ配列とアノテーションの情報が登録されています。 GEO の “Find Platform” のサービスを利用して、自分が使用したマイクロアレイの Platform ID を検索することができます。カスタムのマイクロアレイを使用した場合、Deposit に先立って、その Platform のデータを各自が新規に登録する必要があります。GPLxxxxx という ID で示されます。(xxxxx は数字)
Samples: (さんぷる)
登録されたサンプルは、サンプルごとにIDを割り当てられます。GSMxxxxx というID がそれです。(xxxxx は数字)
Series: (しりーず)
複数の GSMxxxxxx を1セットにしたものが「シリーズ」と呼ばれます。GEOには、最終的にこの1シリーズとして登録され、GSExxxxx というIDが割り当てられます。論文などでサイトする場合は、このシリーズのIDである GSExxxxx を表記します。(xxxxx は数字)
その他、ご不明な点は、お問い合わせください。