マイクロアレイと聞いて、最初に思い浮かぶイメージはどのようなものでしょうか?多くの人が、赤と緑の画像をイメージするでしょう。しかし、この画像は誤解されたイメージかもしれません。
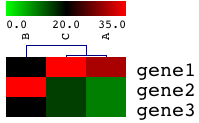
おそらく、イメージは、下の2パターンに別れると思います。

左側をイメージされた方、正解です。これは、マイクロアレイの2色法のイメージです。比較するサンプルを赤と緑の2色でラベルするので、そう呼ばれています(two color, two channelとも)。一方のサンプルで高い発現を示す遺伝子は、赤または緑に見え、両方のサンプルで発現している遺伝子は、赤+緑で黄に見え、両方のサンプルで発現していない遺伝子は黒に見えるという原理です。
しかし、これは古いイメージといえるでしょう。ここ数年の間にマイクロアレイの環境は変わり、近年では1色法が主流です。実際に NCBI のマイクロアレイのデータベースである Gene Expression Omnibus (GEO) に登録されているマイクロアレイデータのほとんどが、 Affymetrix 社の GeneChip® であり、 この GeneChip® は、1色法のマイクロアレイです。また、Agilent 社のマイクロアレイも2色法と1色法の両方に対応しています。
1色でどうやって比較するのか?と疑問を持たれる方もいるかもしれません。端的に言って、マイクロアレイ解析とは画像処理ではありません。解析に用いるのは数値となったデータであり、単純に数字の大小を比較しているに過ぎないのです。考え方としては、1色でも濃淡で比較できると考えていただいてもよいかもしれません。(1色法、2色法それぞれにデメリットとメリットがあります。)
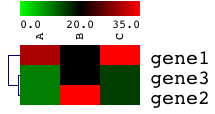
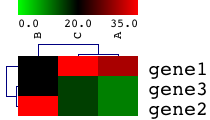
右の画像をイメージされたかたも、ある意味正解です。正確にはヒートマップですが、マイクロアレイデータの表示方法として、論文によく登場するため、このイメージが強いのかもしれません。
しかし、「1色法なのに、どうして、赤と緑の色がついているのだろう?」と思われた方もいるのではないでしょうか。その場合は、本サイトの「MeV の使い方 3. t-検定(続き)」などをご覧ください。シグナル値の色づけは自由です。コントラストが強いため、赤と緑を選ばれることが多いのでしょう。
また、色の意味が異なることにご注意ください。赤=発現が高い、緑=発現が低い、黒=発現していない、ではありません。「黒=発現していない」というのは、上記に述べた2色法の画像での話です。ヒートマップにおいて、「黒=中くらいの値(高くも低くもない)」を意味します。また、ヒートマップの色には「黄(オレンジ)」がないことにもご注意ください。
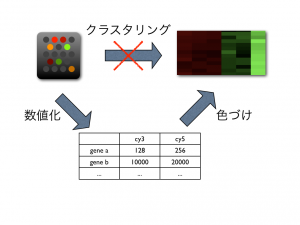
マイクロアレイの画像とヒートマップは、まったく異なるものです。マイクロアレイの画像を整理して並べ替える(クラスタリング処理する)と、ヒートマップが得られるというものではありません。