ダウンロードしたデータを用いて、ボックスプロットを作成してみましょう。マイクロアレイデータは、まず、ボックスプロットや散布図を書いて、シグナル値の状態を確認することをおすすめします。
シグナル値のばらつきが極端に大きい場合は、データのクオリティが良くない(サンプルのコンディションが悪い、RNAの分解が進んでいる)ことも考えられれます。
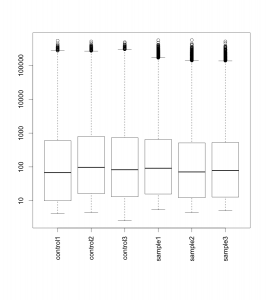
raw データのボックスプロット
raw データ(正規化前)の散布図を示します。便宜上、サンプルの名前を、293T_16hr_Control = control1, 293T_16hr_muTRPV3 = sample1 のように変更しています。サンプルによって、多少上下していることが確認できます。サンプルのクオリティは悪くないように見えます。
使用した raw データの値は、下記よりダウンロードできます。
> https://www.dropbox.com/s/igxolub38mrm0po/rawdata.txt.zip?dl=0
(Agilent の raw データのファイルからシグナル値だけを取り出す方法については割愛します。)
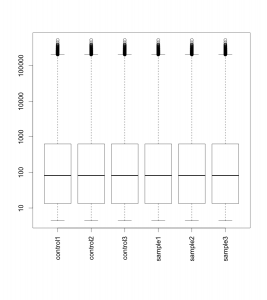
正規化後データのボックスプロット
続いて、正規化後データ (normalized data) のボックスプロットです。ここでは、正規化のアルゴリズムとして、 quantile 法* を用いています。データの分布がそろっていることが確認できます。
使用した正規化後データの値は、下記よりダウンロードできます。
> https://www.dropbox.com/s/2m1poolbqz9vizh/normalized_data.txt.zip?dl=0
極端に分布の異なるサンプルは、正規化の際(アルゴリズムによっては)、他のサンプルのシグナル値にも影響を与えることもあります。物理的なサンプルの状況を確認して、RNAの分解などが疑われる場合は、そのサンプルを除外して正規化を行ったほうがよいでしょう。
* Bolstad et al. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics (2003) vol. 19 (2) pp. 185-93.