ヒストグラム(histogram, 度数分布)
シグナル値の分布に偏りがあるかどうかは、ヒストグラムを書いてみると分かります。ヒストグラムでは、横軸にシグナル値の大きさ、縦軸に一定のシグナル値の区間に存在するプローブ(遺伝子)の数を表します。また、普通の等間隔の目盛では見づらいため、横軸に対数目盛を用います。

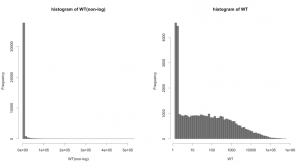
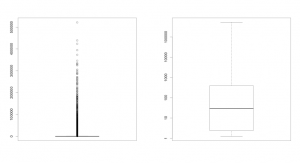
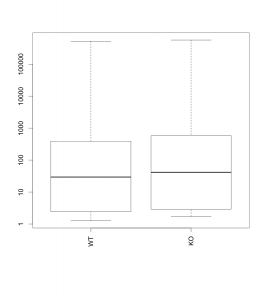
図:シグナル値のヒストグラム:普通の目盛(左)と対数目盛(右)。左の横軸は指数表示になっているが、0, 100000, 200000, 300000, 400000, 500000 ということ。同じデータを使用*[1]。
マイクロアレイデータのヒストグラムは、おおむね図のような形状になります。(メーカーによって、若干の違いはあります。)シグナル値が、かなり低いところに大きな尖った山ができます。また、その右側は比較的なだらかな山が、シグナル値が数十万となるところまで続きます。
尖った山は、この部分に分布している遺伝子の数が極端に多いことを意味します。だいたい、4万個の遺伝子中、約1万個の遺伝子が、この部分に含まれるケースが多いです。ここに含まれる遺伝子のシグナル値は、1桁から2桁程度であり、マイクロアレイにより検出されていない*[2]可能性が高い遺伝子です。シグナル値が低くても、マイクロアレイデータから発現していないと言い切ることはできません*[3]。しかしながら、イメージとして、どのような細胞であっても、その中で1万遺伝子程度は、発現していないというのは生物学的に自然なことではないでしょうか。
右側の比較的なだらかな山は、残りの3万個程度の遺伝子が、数百から数十万までの区間に広く分布していることを意味します。通常、この部分に偏りは少なく、シグナル値の高い区間になるほど、そこに分布する遺伝子の数は、少なくなっていきます。つまり、マイクロアレイでは、高いシグナル値になるほど、稀な値だということです。
ヒストグラムから分かること
シグナル値の分布の偏りが分かります。検出されていない遺伝子の山以外の比較的なだらかな部分に、極端に高い凹凸があれば、データのクオリティが疑われます。
ヒストグラムから分からないこと
特定の遺伝子に発現変動があるかどうかは、ヒストグラムからも判別できません。(それには、散布図を用います。)ヒストグラムは、1サンプルに1つ書けますが、ボックスプロットのように複数のサンプルを並べて同時に比較することには向きません。
ここまでのまとめ
ボックスプロットとヒストグラムから、シグナル値が低い部分に集中している形状を取るのが、マイクロアレイデータの特性と言えます。1桁から数十万まで幅広い値を取ります。また、約4分の1程度は、検出されず、検出された遺伝子も数百程度のシグナル値のものまでが75%を占め、高い値を示すのは残りの1万個程度です。そのため、マイクロアレイデータの解析結果は、これらの事実を考慮した上で、見ていく必要があります。(マイクロアレイは、このようなバイアスを持った測定手法だということが、前提となります。)マイクロアレイデータは、その特性上、発現レベルが低く、微妙な発現変動を示す遺伝子の検出には向かないと考えられます。

[1] GSM830131
[2] 検出されたかどうかは、データにフラグがあれば、その判定を見ます。
[3] シグナル値が低い場合であっても、リアルタイムPCRなど、マイクロアレイより測定精度の高いとされる他の手法では、検出される可能性があります。