GEOへの登録
最近のジャーナルでは、マイクロアレイデータを用いた論文を投稿する際に、マイクロアレイデータの Gene Expression Omnibus (GEO) への登録を求められることが多いです。GEOは、これまでに多くのマイクロアレイデータが登録されており、自由に閲覧が可能なデータベースです。
GEO にマイクロアレイデータを登録すると、”GSExxxxx” という番号をもらうことができます。他の研究者は、この番号を指定することで、そのマイクロアレイデータを閲覧することが可能です。レビューワーから、このアクセッション番号や、IDという言い方で、求められることもあります。
登録する際に準備するもの
GEOにマイクロアレイデータを登録する際に必要なものは、下記の3点です。(ここでは、GEOarchive という形式の登録方法を紹介します。)
- NCBI のアカウント(ユーザー名とパスワード)
- メタデータ(論文の要旨や、サンプルの情報など。)
- マイクロアレイデータ(CELファイルなどのrawデータを含む。)
1. NCBI のアカウント
NCBIを利用するときのアカウントです。NCBIのサイトで無料で作成できます。左下の “Register for an NCBI account” から先へ進みます。(以前は、GEO専用のアカウントがありましたが、現在は、NCBIアカウントに統合されています。)
アカウント作成後、登録されたメールアドレスに、confirm 用のメールが届きます。そのメールに書かれたリンクをクリックすることで、確認されたことになり、アカウントが有効化されます。(迷惑メールと判断されることもあるようですので、ご注意ください。)
2. メタデータ
論文の要旨や、サンプルの情報を専用のエクセルのフォームに記入します。Affymetrix, Agilent, Illumina ごとに専用のフォームがあります。テンプレートのエクセルファイルをダウンロードして記入します。このテンプレートの3枚目以降のシートに記入例があります。各項目の意味はこちらをご覧ください。
3. マイクロアレイデータ
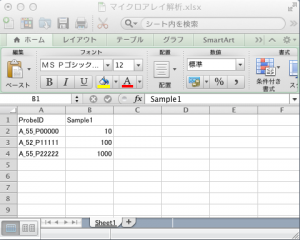
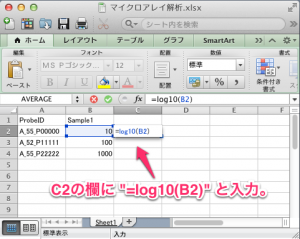
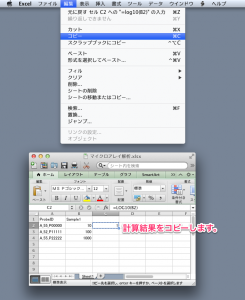
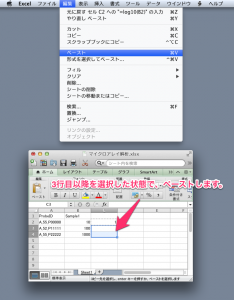
正規化済みのマイクロアレイデータと、これに加えて、rawデータ(AffymetrixであればCELファイル、AgilentであればFeatureExtractionから出力されるファイル)も必要です。ratioやアノテーションの情報は、必須ではありません。プローブIDと、シグナル値と(あれば)フラグの情報を登録します。
正規化に用いたすべてのサンプルの情報が必要です。「8サンプルで正規化を行って解析した後、論文に使用した4サンプルだけを登録」ということはできません。8サンプルで正規化した場合と、4サンプルで正規化した場合では、若干、シグナル値が変わるためです。上記のような場合は、4サンプルだけで再度、正規化から解析しなおして、登録する必要があります。
株式会社セルイノベーターでは、「マイクロアレイデータのGEOへの登録」も受託解析サービスの料金の中に含まれています。登録の際は、ご相談ください。