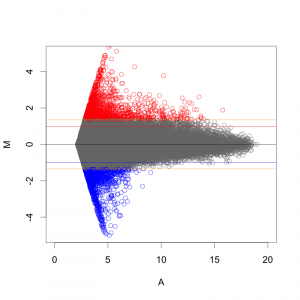
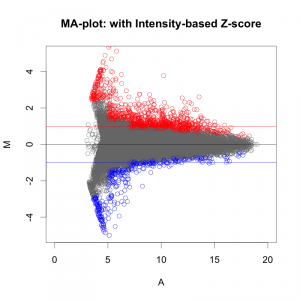
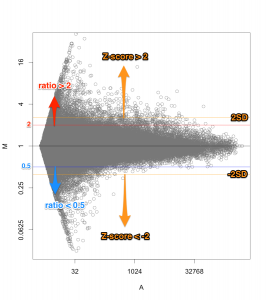
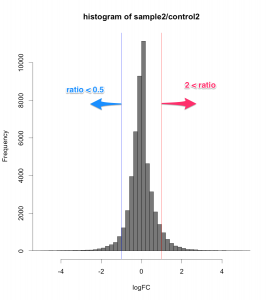
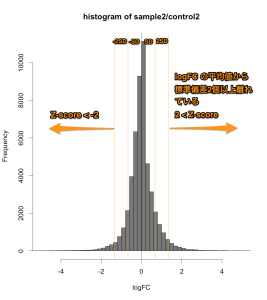
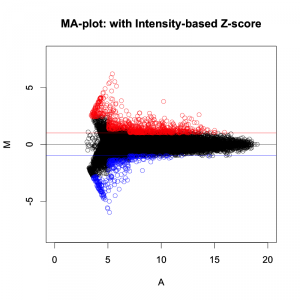
正規化後のシグナル値から、ratio と Z-score を算出して、発現変動を判定します。その結果、発現変動している遺伝子のリスト(例えば、エクセルファイルのようなテーブル)が得られます。ここでは、 sample2/control2 = 24hr どうしを比較したデータを使用しています。
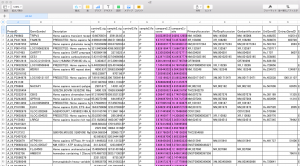
発現変動遺伝子
この例では、下のMAプロットに示されたような部分が変動していると判定されています。判定条件としては、 ratio > 2 かつ Z-score > 2 (または ratio < 0.5 かつ Z-score < -2)という少し厳しめの条件にしています。増加しているもの、減少しているもの合わせて、1032個を変動していると判定しています。
まずは、変動している遺伝子の機能を調べてみましょう。機能を調べるには、データベースの DAVID が便利です。
DAVIDによる機能解析
DAVIDに変動している遺伝子の ID のリストをアップロードすると、そのリストに含まれる遺伝子にどのような機能の遺伝子が多いのか(膜タンパクが多いのか、転写因子が多いのか、アポトーシスに関連する遺伝子が多いのか)、確認することができます。操作方法は、リンク先を参照してください。
DAVID の Functional Annotation Clustering を実行すると、下記のようなテーブルが得られます。
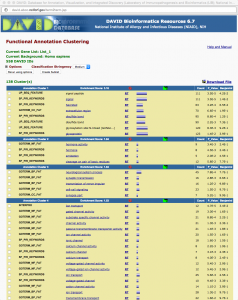
左上の表示を確認すると、1032個の変動遺伝子のうち、558個が認識されたことがわかります。(ここでは、EntrezGeneID をアップロードしています。)変動しているもののうち、アノテーションがついていないもの、機能が未知のものが多かったようです。
テーブルの最初 (Annotation Cluster 1) から、signal peptide, glycoprotein 、つまり、シグナルを伝達する分子、糖タンパクの遺伝子が多かったことが分かります。だいたい、この機能のグループ(クラスター)は、どんなデータでも上位に来ることが多いです。これらの用語は、漠然とした分類のアノテーションなので、あまり、情報としての価値はないかもしれません。
アノテーションのクラスターとして、次は、ホルモンのようですが、遺伝子数はそれほど多くありません。
入手元のデータのサマリーによると、変異の結果、アポトーシスが活性化され、ケラチノサイトの分化が抑制とあります。
TRPV3 dysfunction may increase apoptotic activity, inhibit keratinocyte differentiation and disturb the intricate balance between proliferation and differentiation state of keratinocytes in the skin.
ブラウザの検索機能で、”apoptosis” を検索して見ると、テーブルの下の方に見つかりました。
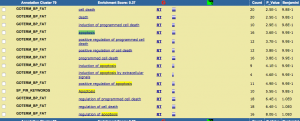
確かに変動しているようですが、数は少ないようです。(1032個のうち、20個程度)Enrichment Score も 0.37 と低いです。
“keratinocyte” を検索すると、 “keratinocyte differentiation” も見つかりますが、これも数は少ないです。

24hr の比較では、あまり、変異体の影響が出ていないように思われます。ほかに 16hr, 40hr のタイムポイントもあるので確認してみる必要がありそうです。