がんゲノムのデータベースとして、 The Cancer Genome Atlas (TCGA) が利用されるようになりました。
TCGA に登録されたデータ
乳癌、肺がん、など、組織ごとに数百サンプルのデータが登録され、公開されています。データには、マイクロアレイをはじめ、メチレーションのデータや、次世代シーケンサーによって取得された配列情報も含まれています。
これらのデータは、ガイドラインを守れば、研究目的として利用が可能です。データをダウンロードして取得し、自分で解析することもできます。(追記:2015年12月末以降、全てのデータセットが公開になりました。詳細は、ガイドラインのページをご確認ください。)
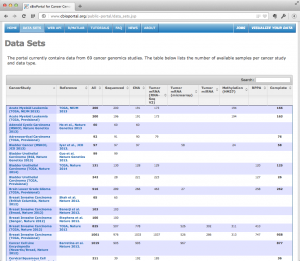
TCGA のデータは、 cBioPortal を通して閲覧可能
イチから解析せずに、データを見てみたいという場合は、 cBioPortal というサイトを通してデータの閲覧が可能です。一部のデータにはなりますが、各がんについて、変異の多い遺伝子などの情報を確認できます。
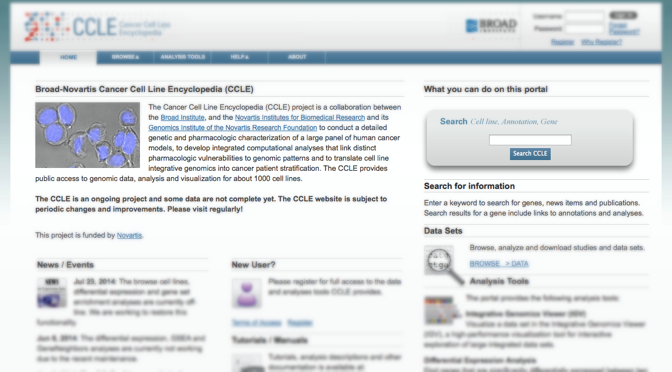
以前紹介した、 Cancer Cell Line Encyclopedia (CCLE) のデータも、 TCGA の一部として扱われています。