インプリンティングを受けることが知られている遺伝子 (imprinted genes) のデータベースがあります。その名の通り、 “geneimprint” です。
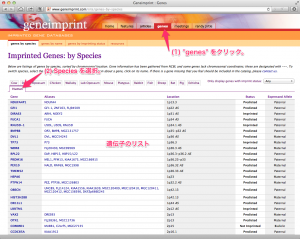
上部の “genes” をクリックすると、インプリンティングを受ける遺伝子のリストが確認できます。生物種ごとにまとめられています。
日本エピジェネティクス研究会のページにも、その他のデータベースへのリンク集があります。
* Dr. Randy L. Jirtle; http://randyjirtle.com
インプリンティングを受けることが知られている遺伝子 (imprinted genes) のデータベースがあります。その名の通り、 “geneimprint” です。

上部の “genes” をクリックすると、インプリンティングを受ける遺伝子のリストが確認できます。生物種ごとにまとめられています。

日本エピジェネティクス研究会のページにも、その他のデータベースへのリンク集があります。
* Dr. Randy L. Jirtle; http://randyjirtle.com
「特定の遺伝子が、どの組織で発現しているか?」を調べるには、 BioGPS を使います。遺伝子名またはタンパク名、プローブID (Affymetrix) で検索すると、対象の遺伝子の複数の組織や細胞における発現レベルが棒グラフで表示されます。また、発現レベルを表示するだけでなく、他のデータベースや、siRNAなどの試薬へのリンクもあります(レイアウトと呼ばれています)。
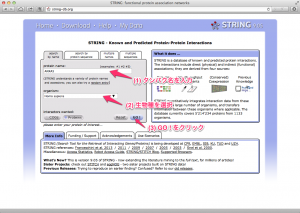
STRING は、タンパク質間相互作用 (Protein-Protein Interaction: PPI) のデータベースです。STRING も GeneMANIA のようにネットワーク図を得ることができます。
使い方も同様です。(1) タンパク名を入力し、(2) 生物種を選択、 (3) GO ! をクリックするだけです。
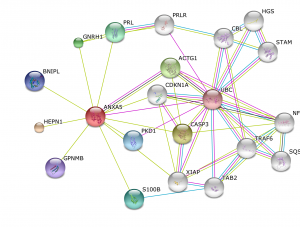
ネットワーク図が表示されます。タンパク質をつなぐ線は、色によって、どの情報由来か示されています。例えば、黄緑色は、Textmining によって得られた関係を示します。
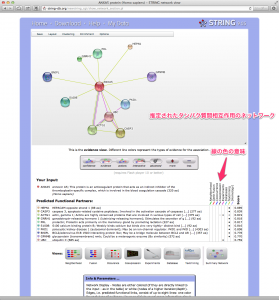
視覚的なネットワーク図を得られますが、GeneMANIAと比べると、ネットワークの情報量は少なめでしょうか。(+のアイコンをクリックして、拡張することもできます。)
* STRING v9.1: protein-protein interaction networks, with increased coverage and integration, Nucl. Acids Res. (1 January 2013) 41 (D1): D808-D815. doi: 10.1093/nar/gks1094
マイクロアレイ解析において、発現変動遺伝子を確認した後、「発現変動していた遺伝子に対するプローブの配列が、ゲノム上のどこに該当するか」を確認したいことがあると思います。これは、 USCS の BLAT で検索することができます。
まず、対象となるプローブの配列情報を入手する必要があります。これは各メーカーによって公開されています。Affymetrix 社であれば、NetAffx から、Agilent 社であれば、 eArray より取得できます。
例えば、Agilent 社のプローブ A_68_P22294038 の配列は、 “ATTCCATTACCATGCAGAACTACTAAAATATATTGTTCAAATAGTAGGAATCTATTTTCT” という60文字です。
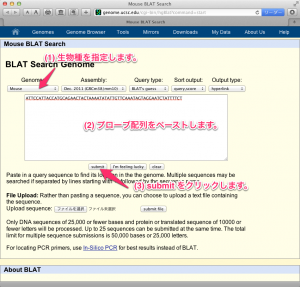
UCSC の BLAT にアクセスします。
http://genome.ucsc.edu/cgi-bin/hgBlat?command=start
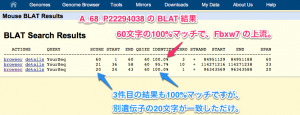
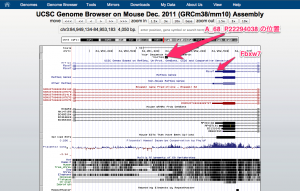
検索結果が表示されます。複数ヒットすることもありますが、Score とマッチ率を参考に正しいものを選びます。ここでは、1件目と3件目が100%マッチですが、3件目はプローブの60文字中、20文字が一致しただけです。左端の “ACTIONS” から “browser” をクリックすると、位置の結果を表示できます。
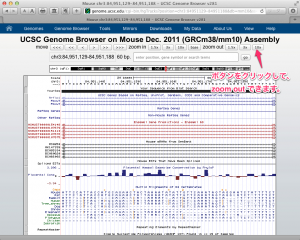
位置が表示されます。ボタンをクリックして、何度か Zoom out すると、プローブの対象となる遺伝子との位置関係を把握しやすくなります。ここでは、”Fbxw7″ が対象の遺伝子です。図中に “Your Sequence” とあるのが、1. で入力したプローブ配列です。1kbほど上流にあることが確認できます。
MSigDB から遺伝子セットを検索して、取得する方法を紹介します。
(1) “Search” をクリックして、遺伝子セットの検索を行います。
(2) メールアドレスを登録(入力)して、ログインします。
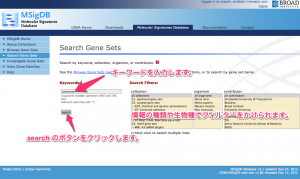
(3) 検索画面が表示されるので、検索したいキーワードを入力し、Search ボタンをクリックします。右側の項目を選択することで、情報の種類や生物種でフィルターをかけることもできます。
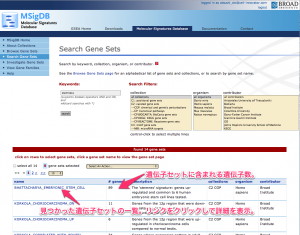
(4) 検索結果に、見つかった遺伝子セットの一覧が表示されます。リンクをクリックすることで、詳細を表示します。#genes に表示されている数字は、遺伝子セットに含まれる遺伝子の個数を意味しています。
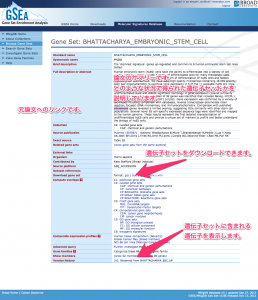
(5) 検索結果の詳細には、元になった論文の情報などが表示されます。なかほどの “Download gene set” の項目から、遺伝子セットをダウンロードできます。遺伝子セットは、複数のフォーマットで用意されています。”text”形式が最もシンプルです。この検索結果の詳細では、画面下の “Show members” の項目をクリックすることで、遺伝子セットを表示して確認することもできます。
なぜか、トップ画面の “Search” から検索した結果と、同画面の “Browse” から検索した結果が異なるようです(2012年12月現在)。 例は、 “stemness” で検索した結果ですが、”Search” から検索した場合がたくさんヒットしました。しかし、 “Browse” から検索しないと表示されない遺伝子セットもありました。