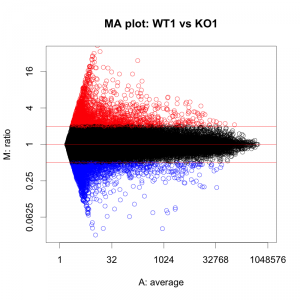
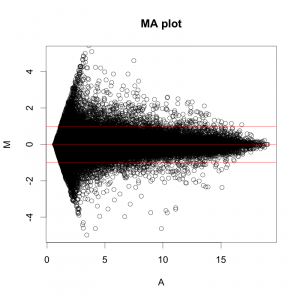
高いシグナルと、低いシグナルがあるためにうまくいかないのであれば、シグナル値を fold change (ratio) に変換して色づけしてはどうでしょうか?
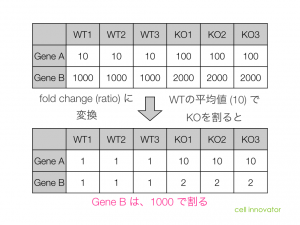
ratio の算出
例の場合、WTをコントロールとするので、WTの平均値で、KOのシグナル値を割ると ratio になります。
Gene A の場合は、WT の平均値が 10 なので、 KO の ratio は、10 になります。一方、WTは同じ値で割るので、1になります。(例は、シグナル値がいずれのサンプルも同じ(=ばらつきが全くない)極端なケースです。)
Gene B の場合は、WT の平均値が 1000 なので、 KO の ratio は、2 になります。WTは同じ値で割るので、1になります。
ratio で色づけ
これを色づけしてみます。変動のないところ、つまり、 ratio = 1 の部分を黒、2倍以上 (ratio > 2) を赤、0.5倍以下 (ratio < 0.5) を緑に塗るとします。
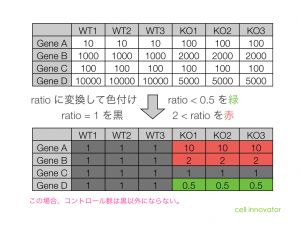
結果としては、コントロール群が黒で、実験群が赤、黒、緑となるヒートマップが得られます。シグナル値が高いところも低いところも、 ratio が変動していれば、変化は色として確認できます。(この意味では、目的を達成している色づけ方法といえます。)
ただ、コントロール群は、同じ値で割ることになるので、黒以外になりません。よって、ratio で色づけした場合、コントロール群をヒートマップに表示する意味はほとんどありません。
ratio を用いた場合、コントロール群で低いところは緑、高いところは赤(実験群はその逆)という、色の変化にはなりません。