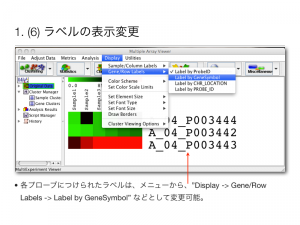
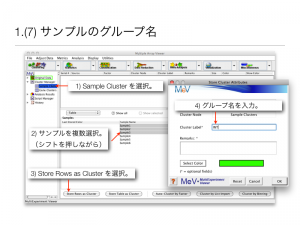
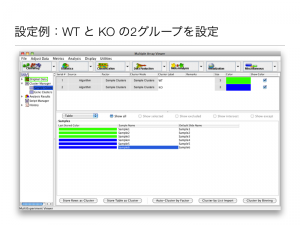
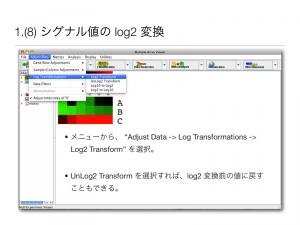
Agilent のマイクロアレイデータを想定して、 MeV の操作方法を紹介します。
MeV の起動とファイルの読み込み
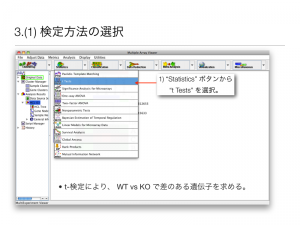
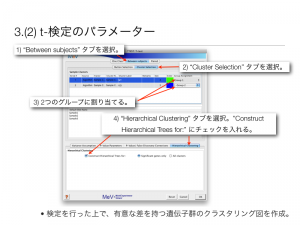
階層的クラスタリング t-検定
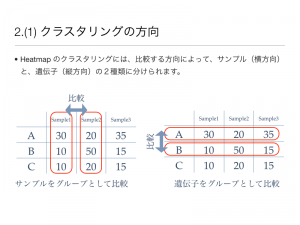
2. 階層的クラスタリング (1) クラスタリングの方向
ヒートマップのクラスタリングには、比較する方向によって、サンプル(横方向) と、遺伝子(縦方向)の2種類に分けられます。どちらの方向でクラスタリングを行うかは、見たいものによって異なります。似ているサンプルを知りたいときは、サンプル方向でクラスタリングを行います。(がん細胞と正常細胞のデータを比較する場合など。)また、発現パターンの似ている遺伝子を知りたい場合は、遺伝子方向でクラスタリングを行います。(特定の遺伝子に興味があり、その遺伝子と発現パターンの似ている遺伝子を探す場合。)
場合によっては、サンプル、遺伝子の両方向で行うこともあるかもしれません。ただ、サンプルが時系列に並んだタイムコースのデータの場合は、サンプル方向のクラスタリングは行わない方が、発現パターンが分かりやすいと思います。
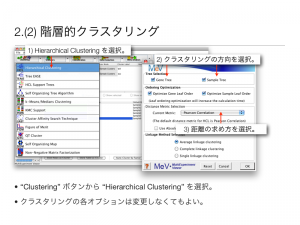
クラスタリングの方向 (2) 階層的クラスタリング
MeV で階層的クラスタリングを行う方法です。
“Clustering” ボタンから “Hierarchical Clustering” を選択。
クラスタリングの各オプションは変更しなくてもよい。
階層的クラスタリング
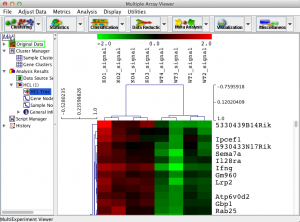
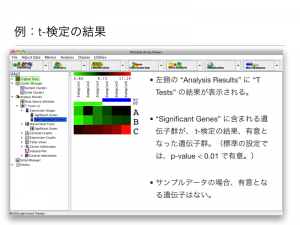
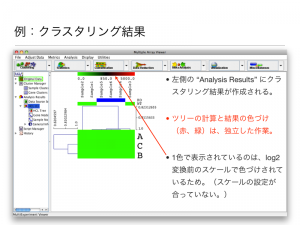
例:クラスタリング結果
左側の “Analysis Results” にクラスタリング結果が作成される。
ツリーの計算と結果の色づけ (赤、緑)は、独立した作業。 1色で表示されているのは、log2 変換前のスケールで色づけされているため。(スケールの設定が合っていない。)
例:クラスタリング結果
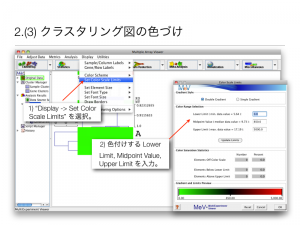
(3) クラスタリング図の色づけ
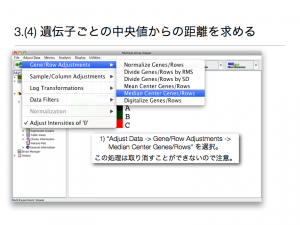
クラスタリング図を適切な表示にするには、「色づけ」の作業が必要です。論文などではシグナル値の低い遺伝子を緑色 、中間の遺伝子を黒色 、高い遺伝子を赤色 に色付けされることが多いです。しかしながら、シグナル値がどれくらい低ければ、緑色 、どれくらい高ければ赤色 という決まりはありません。よって、作者が色付けを定義しなければなりません。MeV では、下記の操作で色づけを定義できます。
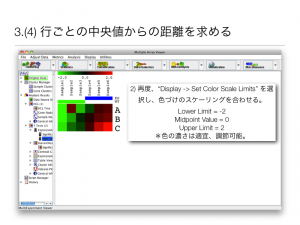
1) “Display -> Set Color Scale Limits” を選択。
2) 色付けする Lower Limit, Midpoint Value, Upper Limit を入力
追記:Color Scale Limits ウィンドウ内の Color Range Selection には、 ()内に参考までの値が表示されています。Lower Limit に「最小値」、Midoint Value に「中央値」、Upper Limit に「最大値」が表示されますが、右側のボックスへの入力自体は、ユーザーが行わなければなりません。
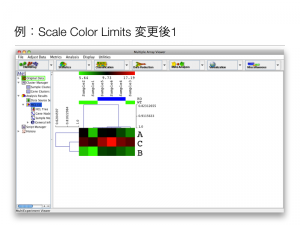
クラスタリング図の色づけ 例: 色づけを変更した後のクラスタリング図
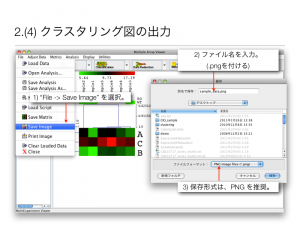
色づけを変更した後のクラスタリング図 (4) クラスタリング図の出力
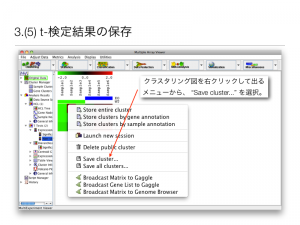
作成したクラスタリング図は、画像ファイルとして出力できます。
1) “File -> Save Image” を選択
2) ファイル名を入力。 (.pngを付ける)
3) 保存形式は、PNG を推奨。
クラスタリング図の出力