GEO に公開されたデータを利用して、実際に解析を行ってみましょう。マイクロアレイ解析によって、どのような情報が得られるのかイメージがつかめると思います。
マイクロアレイデータ
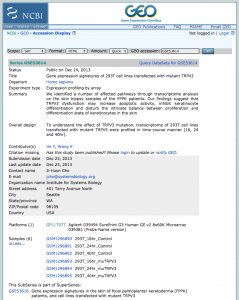
まずは、 GSE53614 のデータを使ってみます。293T cell lines を用いた解析のようです。サンプル名から推測するに、3つのタイムポイント (16hr, 24hr, 40hr) があり、それぞれに、コントロールと対照サンプルがあるようです。また、使用されたアレイは、 Agilent SurePrint G3 Human GE 8x60K の v2 です。
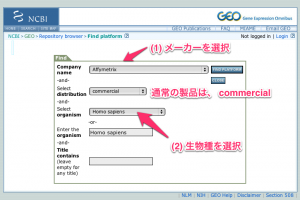
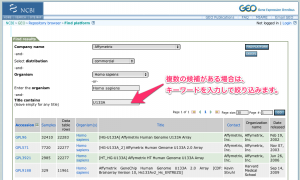
マイクロアレイデータの取得
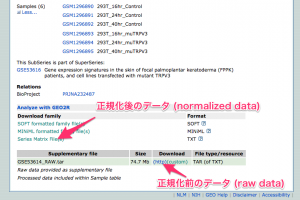
データを取得します。取得する方法は、いくつかあります。著者らが正規化したデータをそのまま利用するには、 “Series Matrix Files(s)” をダウンロードしましょう。正規化からやり直す場合は、その下の GSE53614_RAW.tar をダウンロードします。 (ここでは、正規化前のデータを用いました。)
なお、 SOFT形式のデータをダウンロードすれば、アノテーションが付いた状態のファイルを取得できますが、計算時に読み込むときに苦労する場合があります。また、MINiML形式の場合も、XML形式のデータであるため、計算に用いない情報が含まれています。
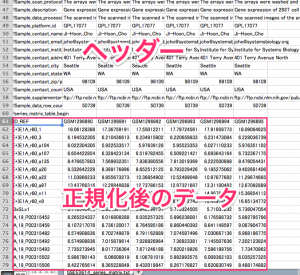
Series Matrix File の始めの行(ヘッダー)は、サンプル情報です。 ID_REF から始まる行以降のデータが、正規化後のデータです。1列目はプローブのIDです。2列目以降に各サンプルのシグナル値があります。(6サンプルなので6列)プローブの ID が A_ で始まらないものは、バックグラウンドなどの値です。