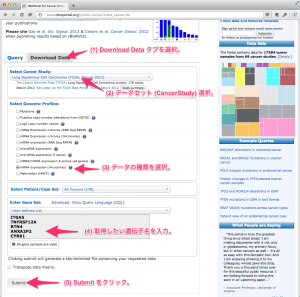
iPS 細胞のデータを正規化したら、何はともあれ、 stem cell 関連遺伝子の発現はどうなっているのかが気になるところだと思います。
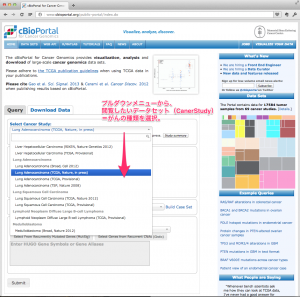
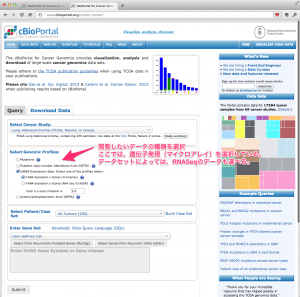
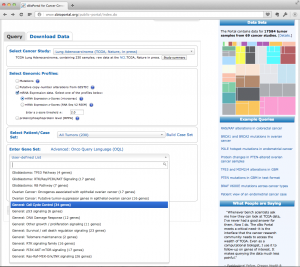
このようなときは、アノテーションを元に、 stem cell 関連遺伝子だけを抽出して、クラスタリング処理し、さらにヒートマップで表示するとよいでしょう。(この段階では、ratio や p-value を算出してないので、変動していないものも含めています。)
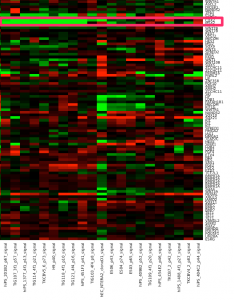
stem cell 関連遺伝子のヒートマップ
作成したヒートマップの結果を示します。BioGPSのデータを用いたヒートマップでは、 ES細胞に特徴的な遺伝子発現のパターンが見えていましたが、今回のデータではどうでしょうか?

とりあえず、大きく分けると3つのクラスターに分かれているように見えます(あくまで見えているというだけで、以下、決定的な事実というわけではありませんので、注意してください)。
一番左のクラスターには、HepG2 や MCF7 などのがん細胞が含まれています。どうやら、stem cell 関連遺伝子に関しては、 iPS細胞とがん細胞で似ていないようです。また、iPS細胞では、がん細胞ほど共通のパターンが見られません。いろいろなバリエーションのiPS細胞がありそうです。
上部のツリーの分かれ方を見ると、iPS 細胞は、さらに大きく2つのグループに分かれるようです。この2つのグループで、はっきり分かれているように見える遺伝子としては、上から3分の1ほどのクラスターに含まれる PSCA, GATA2, CDX2, MSX2, HOXB4 あたりでしょうか。
ヒートマップから読み取れる情報
ヒートマップの色付けは、 median からの距離にしていますので、 色が黒いところが低いというわけではありません。特に、一番下の行のクラスターに含まれている POU5F1 (OCT3) や NANOG は、黒く見えていますが、左側のがん細胞のデータが明るい緑なので、がん細胞よりiPSにおける発現は高いことを意味します。
また、1つのサンプルの遺伝子を1つ取って見ると、それが MCF7 に近いほど赤であったり、緑であったりします。個別に見ることには、あまり意味がないかもしれません。例えば、hiPS201B2_p67 の MSX2 や CDX2 の色は、HepG2と同程度の緑色ですが、これだけを持って何か結論づけることはできません。
繰り返しになりますが、色ではなく、実際の値を確認するほうがよいです。シグナル値が1桁程度のものでも、このような色付け方法では、差が強調されすぎる場合があります。
がん細胞だけではなく、BioGPSのように、いろいろな分化後の組織が混ざっていると、色のパターンが出やすかったかもしれません。(iPS細胞はがん細胞とは違う、ということを確認するという意味ではこれだけでよかったのかもしれませんが。)