MeV を用いて、ペアを考慮した t-検定を行う方法を紹介します。
検定を行う2グループに含まれるサンプルの間に、ペアの関係がある場合は、ペアを考慮した検定が可能です。(例えば、患者ごとに投与前、投与後のサンプルある場合など。)ここでは、仮に WT と KO にペアの関係があったと仮定して、例として用いています。
MeV を用いて、サイズの大きなヒートマップを作成した場合、結果の画像をファイルとして保存できないことがあります。
例えば、5000個の遺伝子でヒートマップを作成した場合、Set Element Size で指定する Element Width と Height を 40 x 20 くらいにしていると、この問題が発生します。
1つの対処方法としては、 Element Width と Height を 40 x 1 のようにして、縮小されたヒートマップにする方法があります。ただ、この場合、保存したヒートマップは小さすぎて、個々の遺伝子名は読めません。
数千遺伝子のヒートマップを、文字が読める大きさで画像ファイルに保存するには、MeV の利用できるメモリを増やすことで対応できるときがあります。ここでのメモリは、パソコンに搭載されたメモリのことではありません。初期状態で、パソコンに搭載されたメモリのうち、MeVの利用できるメモリは、1GBに制限されています。この制限を変更する方法を示します。(Macの場合です。)
まず、テキストファイルを編集するためのソフト(テキストエディタ)を準備してください。 “Text Wrangler” がおすすめです。必要な場合は、App Store からダウンロードしてインストールします。
次に MeV のフォルダを開きます。MeVをインストールした場所にあります。通常は、「アプリケーション」フォルダにあると思います。
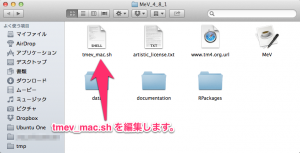
そのフォルダに含まれる “tmev_mac.sh” というファイルを “TextWrangler” などのテキストエディタで編集します。 “tmev_mac.sh” をクリックして、右クリックもしくは、上部のメニューから、「このアプリケーションで開く」–> 「TextWrangler」を選択します。
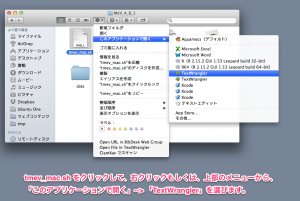
最終行の “1024” を “4096” などの大きい数字に変更します。ここは、パソコンに実際に搭載されたメモリの大きさと相談してください。搭載されたメモリの半分ほどは、MeVに割り当てても問題ありません。(例:メモリを8GB搭載している場合は、4GB = “4096” を指定。もし、2GB割り当てるのなら “2048” などとします。)
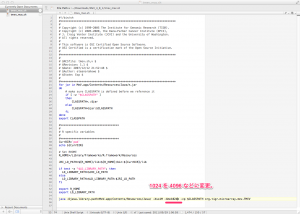
編集後は、下記のようになります。ファイルを上書き保存してください。(この設定ファイルの編集は、インストールされたMeVへの影響はありませんので、この変更により、通常の MeV が起動できくなることはありません。)

編集したファイルから、MeV を起動すると、メモリの制限を変更した状態で MeV を利用できます。TextWrangler のメニューの「#!」から、「Run in Terminal」を選択してください。
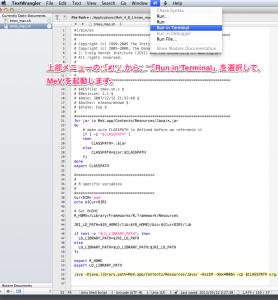
なお、tmev_mac.sh を上書き保存したとしても、変更したMeVを利用するには、毎回、このファイル経由で MeV を起動する必要があります。(MeV のアイコンをダブルクリックした場合は、標準状態の MeV が起動します。)
MeV でヒートマップを作成した際に、保存した画像ファイルの右側に大きく余白が生じてしまうことがないでしょうか?これは画像のサイズが、MeVのウィンドウの大きさの影響を受けているために生じた余白です。
対処方法としては、あらかじめ、ウィンドウの大きさを小さくしてから(全体が表示されない程度に)、画像の保存を行います。
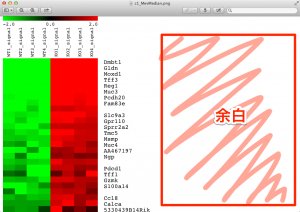
対処方法:ウィンドウサイズを小さくします。MeVのウィンドウの右下をドラッグして、サイズを調節します。右側に余白が表示されないようにウィンドウの幅を調節してください。
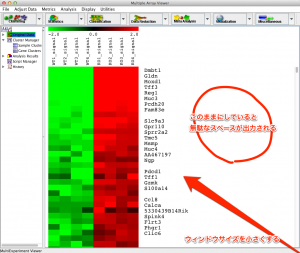
ヒートマップの画像の全体が表示されない程度に、ウィンドウを小さくしてから、 File –> Save Image を選んで、ヒートマップを保存します。
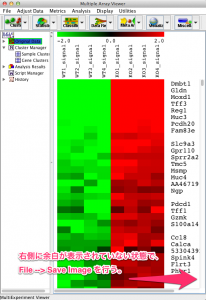
MeV でクラスタリング図を作成した際に、ツリーの左右が入れ替わってほしいことがあります。例えば、下図のような結果になったとして、左側に緑色(減少している遺伝子)、右側に赤色(増加している遺伝子)になっていたほうがよかったとします。
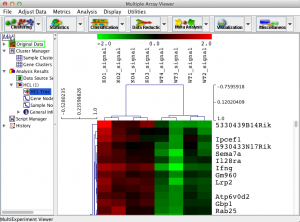
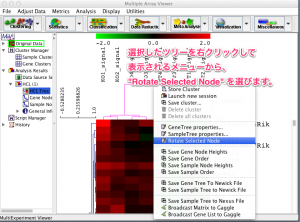
(1) 表示を入れ替えるには、まず、入れ替えたいツリーをクリックして選択します。ツリーの選択された部分は、ピンクにハイライトされます。
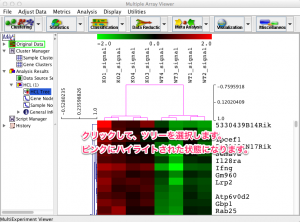
(2) ツリーが選択された状態で、ツリーを右クリックすると、メニューが表示されます。そのメニューから、 “Rotate Selected Node” を選びます。
これで表示が反転された結果が得られます。
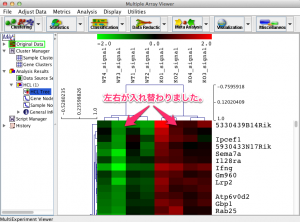
ただし、あくまで見た目の問題であることにご注意ください。ツリーの計算結果をコントロールする処理ではありません。
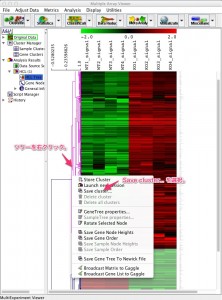
MeV では、クラスタリングした結果から、任意のクラスターを選択し、その選択されたクラスターに含まれる遺伝子群をリストとして出力できます。
クラスタリングの結果を表示したヒートマップ上で、任意のクラスターの選択が可能です。ここでは、WTで低く、KOで高い遺伝子群を含むクラスターを選択する例を示します。
ヒートマップの左側には、クラスターの関係を表したツリーが表示されています。このツリーをクリックすることで、任意のクラスターの選択を行います。クリックするツリーの場所(分岐点)により、選択されるクラスターが決定されます。選択されたツリーは、ハイライトされます。また、選択されなかったクラスターは、表示が薄くなります。
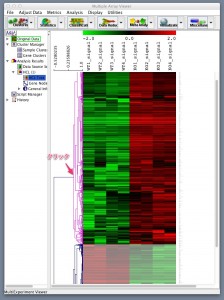
クラスターを選択した状態で、ツリーを「右クリック」し、表示されるメニューから、「Save cluster…」を選択することで、その選択されたクラスターに含まれる遺伝子群をリストとして出力できます。出力されるファイルは、タブ区切りのテキストファイルの形式です。保存するファイルの名前に拡張子(.txt)を含めるようにしてください。出力された遺伝子リストに含まれるシグナル値の値は、MeV で処理された後の値になります。ここでは、ヒートマップ用の色づけを行っているため、log2変換し、中央値からの距離に変換された値となります。