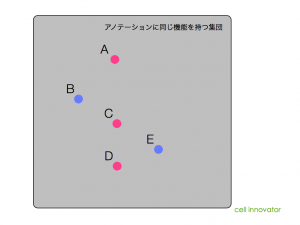
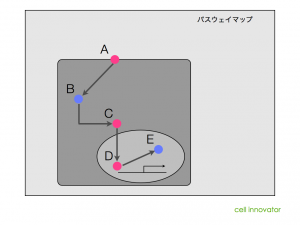
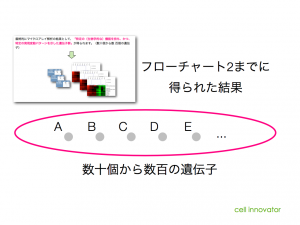
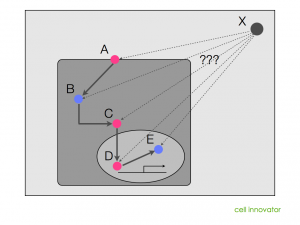
前に、「上流解析には、パスウェイ解析やネットワーク解析を用いる」と述べましたが、どんなパスウェイやネットワーク図であっても、上流解析ができるわけではありません。パスウェイやネットワークの図に含まれる情報には制約があります。「図の中に含まれる遺伝子、を制御している可能性のある遺伝子X」が載っていないのであれば、上流解析はできません。
例えば、KEGGに代表されるような代謝経路の図(マップ)は、代謝される物質と、それを触媒する酵素についてまとめられたマップです。そのため、ほとんどの場合、それらの酵素を制御する遺伝子についての言及はありません。
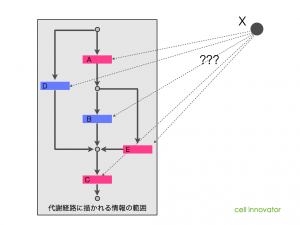
同様に、TGF-beta のパスウェイなど、既知のシグナル伝達系についてまとめられたパスウェイ(カノニカルパスウェイなどと呼ばれます)についても同様です。多くの場合、「シグナルの流れ」に注目してまとめられているため、「シグナル伝達を行う遺伝子、を制御する遺伝子」は、載っていません。
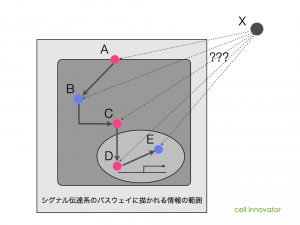
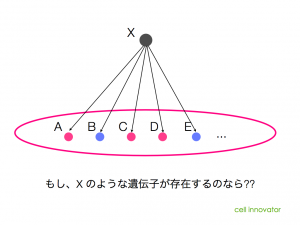
では、どうやって、Xを探せばよいのでしょうか?
続きます。