「遺伝子セット」のデータベースが、 Molecular Signatures Database (MSigDB) です。Broad institute の GSEA 内にあります。メールアドレスを登録することで閲覧が可能です。
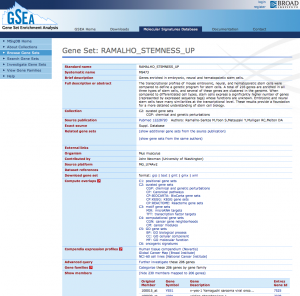
遺伝子セット
MSigDB の遺伝子セットは、大きく分けて6つのコレクションから構成されています。
- c1: positional gene sets, 染色体の座標によるもの。
- c2: curated gene sets, キュレーターが論文から取り出したもの。
- c3: motif gene sets, 転写制御のモチーフごとのリスト。
- c4: computational gene sets: がん由来のマイクロアレイデータをコンピューターで分析して作成したリスト
- c5: GO gene sets, Gene Ontology (GO) から作成したリスト。
- c6: oncogenic signatures, さまざまな因子の影響下にある、がん細胞のマイクロアレイデータから作成したリスト。
MSigDB の代表的な遺伝子セットとしては、c2 の論文から取得されたリストでしょう。論文に書かれた遺伝子群をキュレーターがチェックして、遺伝子セットとして登録しています。また、この中には、BioCarta, KEGG, Reactome など、パスウェイに関するリストも含まれています。また、 c5 には、GO から得られた遺伝子セットもあります。つまり、GSEA を行うと、パスウェイ解析もGO解析も同時に行えるといえます。(パスウェイの色づけはなく、アノテーションが最新とは限らないため、完全な代用にはなりませんが。)
> Subramanian, Tamayo, et al. (2005, PNAS 102, 15545-15550)