Agilent のマイクロアレイデータを想定して、 MeV の操作方法を紹介します。
- MeV の起動とファイルの読み込み
- 階層的クラスタリング
- t-検定
3. t-検定
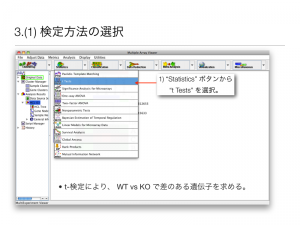
t-検定により、WT vs KO で差のある遺伝子を求める。
(1) 検定方法の選択
- “Statistics” ボタンから “t Tests” を選択。
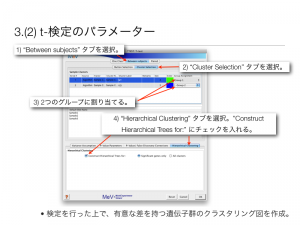
(2) t-検定のパラメーター
検定を行った上で、有意な差を持つ遺伝子群のクラスタリング図を作成。
- 1) “Between subjects” タブを選択。
- 2) “Cluster Selection” タブを選択。
- 3) 2つのグループに割り当てる。
- 4) “Hierarchical Clustering” タブを選択。”Construct Hierarchical Trees for:” にチェックを入れる。
続きます。

下記の仮想データを例に用いています。
ProbeID GeneSymbol Sample1 Sample2 Sample3 Sample4 Sample5 Sample6
A_04_P003444 A 100 1000 200 400 800 900
A_04_P003442 B 50 100 200 300 400 450
A_04_P003443 C 2000 4000 5000 10000 20000 150000