クラスタリングを解釈する際に、よく誤解しやすい部分があります。クラスタリングの結果というものは、それほど絶対的なものではありません。
クラスタリングの結果は、どのアルゴリズム(hierarchical, k-means, SOM など)を用いてクラスタリングするかで多少異なりますし、同じアルゴリズムを用いたとしても、パラメーターの設定によっても若干変化します。
また、クラスタリングするときに、全遺伝子を用いるか、変動している遺伝子だけを用いるかでも変わります。
サンプル間の距離の計算方法による違い(パラメーターの違い)
サンプル方向にクラスタリングを行い、似ている順位のツリーを書くためには、各サンプル間の距離を求めることになります。このサンプル間の距離を求める方法は、いくつかあります。例えば、単純に引き算して差を取るだけの場合や、2乗してから差を取る場合、相関係数を用いる場合などです。
MeV では、 Distance Metric Selection の部分で、この距離を求める手法を選択することになります。
このサンプル間の距離の求め方によって、最終的な結果のツリーの分類は多少異なります。解析例1の全遺伝子(5万プローブ)をサンプル方向のみでクラスタリングした結果を示します。
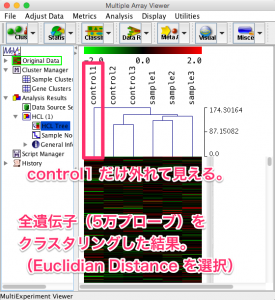
距離を求める手法に Euclidian Distance を選択した場合、control1 だけ外れて(似ていないように)見えます。
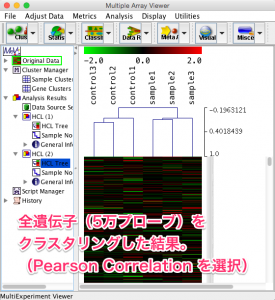
相関係数(Pearson Correlation)を用いて、クラスタリングした場合、control1だけ外れているようには見えません。一般的には、この相関係数を用いることが多いと思います。
クラスタリングに用いる遺伝子セットによる違い
上述の全遺伝子を用いてクラスタリングした結果に対して、変動している遺伝子だけをクラスタリングした結果を示します。 全遺伝子を対象とした場合と異なり、 sample2 と sample3 が近いように見えています。

このようにクラスタリングの結果は、必ずしも、絶対的なものではありません。よく論文で図示されていますが、あくまで1例であり、あるパターンに分かれる可能性を示したものにすぎません。(仮説の証明手段ではありません。)
特に全遺伝子でクラスタリングした場合、「sample1 と一番近いのは、 sample2 と sample3 のどちらか?」というような、結果の細部にあまりこだわらないほうがよいでしょう。前述のような計算方法の違いで多少前後する可能性があります。ある意味、当然のことなので、これらの結果に一喜一憂する必要はありません。
(その時、用いたデータセットとパラメーターを使えば、とりあえず、あるパターンに分かれて見えるという程度に理解した方が良いです。結果の解釈には生物学的な裏付けが求められます。)
