「ある特定の遺伝子が、どのパスウェイに載っているのか?」知りたいことがあると思います。この場合、 NCBI で検索することで、簡単にパスウェイの一覧を得ることができます。
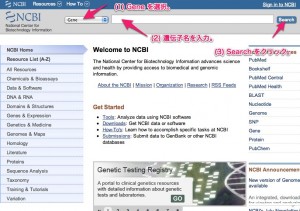
1. NCBI で検索
(1) NCBI のサイトで、対象に “Gene” を選択します。(2) 目的となる遺伝子の名前(Official Gene Symbol など)を入力します。(3) Search をクリックします。
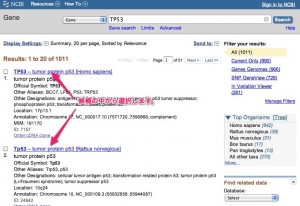
2. 検索結果
検索した遺伝子の候補が表示されます。生物種などを手がかりに、目的の遺伝子を選択します。
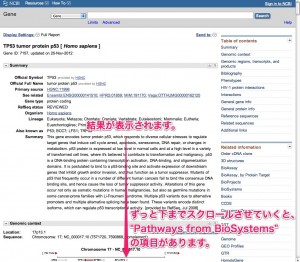
3. 遺伝子の詳細な情報
目的となる遺伝子の詳細な情報が表示されます。さまざまな情報を確認できますが、画面をずっと下までスクロールさせていくと、”Pathways from BioSystems” という項目があります。
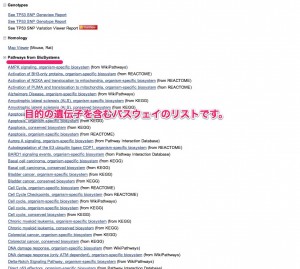
4. Pathways from BioSystems
パスウェイの一覧が見つかります。パスウェイは、各パスウェイデータベースの情報とリンクしています。同じ名前のパスウェイであっても、元となるパスウェイデータベースによって中身が少しずつ異なっています。また、パスウェイとして扱われていても、パスウェイのマップ(ダイアグラム、絵)が描かれていないものも多数あります。KEGGパスウェイデータベースのマップが一番参考になると思います。